適合度檢定#
- scipy.stats.goodness_of_fit(dist, data, *, known_params=None, fit_params=None, guessed_params=None, statistic='ad', n_mc_samples=9999, rng=None)[source]#
執行適合度檢定,比較資料與分布族群。
給定一個分布族群和資料,執行虛無假設檢定,檢定資料是否來自該族群的分布。可以指定分布的任何已知參數。分布的剩餘參數將會擬合到資料中,並相應地計算檢定的 p 值。有多種統計量可用於比較分布與資料。
- 參數:
- dist
scipy.stats.rv_continuous 代表虛無假設下分布族群的物件。
- data1D 類陣列
有限、未刪剪的資料,用於檢定。
- known_params字典,選用
一個字典,包含已知分布參數的名稱-值對。蒙地卡羅樣本是從虛無假設分布中隨機抽取的,並使用這些參數值。在評估觀察到的 data 和每個蒙地卡羅樣本的統計量之前,只將虛無假設分布族群的剩餘未知參數擬合到樣本;已知參數保持固定。如果分布族群的所有參數都是已知的,則省略將分布族群擬合到每個樣本的步驟。
- fit_params字典,選用
一個字典,包含已擬合到資料的分布參數的名稱-值對,例如使用
scipy.stats.fit或 dist 的fit方法。蒙地卡羅樣本是從虛無假設分布中抽取的,並使用這些指定的參數值。但是,在評估統計量之前,這些參數和虛無假設分布族群的所有其他未知參數始終會擬合到樣本,無論是觀察到的 data 還是蒙地卡羅樣本。- guessed_params字典,選用
一個字典,包含已猜測的分布參數的名稱-值對。這些參數始終被視為自由參數,並且擬合到提供的 data 以及從虛無假設分布中抽取的蒙地卡羅樣本。這些 guessed_params 的目的是用作數值擬合程序的初始值。
- statistic{“ad”, “ks”, “cvm”, “filliben”} 或可呼叫物件,選用
用於在將分布族群的未知參數擬合到資料後,比較資料與分布的統計量。可使用 Anderson-Darling (“ad”) [1]、Kolmogorov-Smirnov (“ks”) [1]、Cramer-von Mises (“cvm”) [1] 和 Filliben (“filliben”) [7] 統計量。或者,可以提供簽名為
(dist, data, axis)的可呼叫物件來計算統計量。這裡dist是一個凍結的分布物件(可能帶有陣列參數),data是一個蒙地卡羅樣本陣列(形狀相容),而axis是data的軸,沿著該軸必須計算統計量。- n_mc_samples整數,預設值:9999
從虛無假設分布中抽取的蒙地卡羅樣本數,用於形成統計量的虛無分布。每個樣本的大小與給定的 data 相同。
- rng{None, 整數,
numpy.random.Generator}, 選用 如果 rng 是透過關鍵字傳遞的,則
numpy.random.Generator以外的類型會傳遞給numpy.random.default_rng以實例化Generator。如果 rng 已經是Generator實例,則使用提供的實例。指定 rng 以獲得可重複的函數行為。如果此引數是透過位置傳遞的,或者 random_state 是透過關鍵字傳遞的,則引數 random_state 的舊版行為適用
如果 random_state 為 None(或
numpy.random),則使用numpy.random.RandomState單例。如果 random_state 是整數,則使用新的
RandomState實例,並以 random_state 作為種子。如果 random_state 已經是
Generator或RandomState實例,則使用該實例。
在版本 1.15.0 中變更:作為從使用
numpy.random.RandomState過渡到numpy.random.Generator的 SPEC-0007 轉換的一部分,此關鍵字已從 random_state 變更為 rng。在過渡期間,這兩個關鍵字將繼續有效,但一次只能指定一個。在過渡期之後,使用 random_state 關鍵字的函數呼叫將會發出警告。random_state 和 rng 的行為如上所述,但在新程式碼中應僅使用 rng 關鍵字。
- dist
- 回傳值:
- resGoodnessOfFitResult
具有以下屬性的物件。
- fit_result
FitResult 代表提供的 dist 擬合到 data 的物件。此物件包含分布族群參數的值,這些值完全定義了虛無假設分布,也就是從中抽取蒙地卡羅樣本的分布。
- statistic浮點數
比較提供的 data 與虛無假設分布的統計量值。
- pvalue浮點數
虛無分布中元素的比例,其統計量值至少與提供的 data 的統計量值一樣極端。
- null_distributionndarray
從虛無假設分布中抽取的每個蒙地卡羅樣本的統計量值。
- fit_result
註解
這是一種廣義的蒙地卡羅適合度檢定程序,其特殊情況對應於各種 Anderson-Darling 檢定、Lilliefors 檢定等。該檢定在 [2]、[3] 和 [4] 中被描述為參數式自助法檢定。這是一種蒙地卡羅檢定,其中指定樣本抽取分布的參數是從資料中估計出來的。我們在全文中使用「蒙地卡羅」而不是「參數式自助法」來描述檢定,以避免與更熟悉的非參數式自助法混淆,並在下面描述如何執行檢定。
傳統的適合度檢定
傳統上,對應於一組固定的顯著性水準的臨界值是使用蒙地卡羅方法預先計算的。使用者通過僅計算觀察到的 data 的檢定統計量值,並將此值與製表的臨界值進行比較來執行檢定。這種做法不是很靈活,因為並非所有分布以及已知和未知參數值的組合都有表可用。此外,當從有限的製表資料中內插臨界值以對應使用者的樣本大小和擬合參數值時,結果可能不準確。為了克服這些缺點,此函數允許使用者執行適用於其特定資料的蒙地卡羅試驗。
演算法概述
簡而言之,此常式執行以下步驟
將未知參數擬合到給定的 data,從而形成「虛無假設」分布,並計算此資料和分布對的統計量。
從此虛無假設分布中抽取隨機樣本。
將未知參數擬合到每個隨機樣本。
計算每個樣本與已擬合到樣本的分布之間的統計量。
將來自 (1) 的 data 對應的統計量值與來自 (4) 的隨機樣本對應的統計量值進行比較。p 值是統計量值大於或等於觀察到的資料的統計量的樣本比例。
更詳細地說,步驟如下。
首先,使用最大概似估計將 dist 指定的分布族群的任何未知參數擬合到提供的 data。(一個例外是位置和尺度未知的常態分布:我們使用偏差校正的標準差
np.std(data, ddof=1)作為尺度,如 [1] 中建議的那樣。)這些參數值指定了分布族群的特定成員,稱為「虛無假設分布」,也就是在虛無假設下從中抽取資料的分布。statistic(它比較資料與分布)是在 data 和虛無假設分布之間計算的。接下來,從虛無假設分布中抽取許多(特別是 n_mc_samples 個)新樣本,每個樣本包含與 data 相同數量的觀察值。分布族群 dist 的所有未知參數都擬合到每個重採樣,並且在每個樣本及其對應的擬合分布之間計算 statistic。這些統計量值形成了蒙地卡羅虛無分布(不要與上面的「虛無假設分布」混淆)。
檢定的 p 值是蒙地卡羅虛無分布中統計量值的比例,這些值至少與提供的 data 的統計量值一樣極端。更精確地說,p 值由下式給出
\[p = \frac{b + 1} {m + 1}\]其中 \(b\) 是蒙地卡羅虛無分布中統計量值大於或等於為 data 計算的統計量值的數量,而 \(m\) 是蒙地卡羅虛無分布中元素的數量(n_mc_samples)。分子和分母加 \(1\) 可以認為是在虛無分布中包含了與 data 對應的統計量值,但在 [5] 中給出了更正式的解釋。
限制
對於某些分布族群,檢定可能非常緩慢,因為分布族群的未知參數必須擬合到每個蒙地卡羅樣本,並且對於 SciPy 中的大多數分布,分布擬合是通過數值最佳化執行的。
反模式
因此,可能會很想將預先擬合到 data(由使用者)的分布參數視為 known_params,因為指定分布的所有參數排除了將分布擬合到每個蒙地卡羅樣本的需要。(這基本上是原始 Kilmogorov-Smirnov 檢定的執行方式。)雖然這樣的檢定可以提供反對虛無假設的證據,但該檢定是保守的,因為小的 p 值往往會(大大)高估犯第一型錯誤的機率(也就是說,即使虛無假設為真也拒絕它),並且檢定的檢定力很低(也就是說,即使虛無假設為假,也不太可能拒絕虛無假設)。這是因為蒙地卡羅樣本不太可能像 data 一樣與虛無假設分布一致。這往往會增加虛無分布中記錄的統計量值,因此更多的值超過 data 的統計量值,從而誇大 p 值。
參考文獻
[1] (1,2,3,4,5)M. A. Stephens (1974). “EDF Statistics for Goodness of Fit and Some Comparisons.” Journal of the American Statistical Association, Vol. 69, pp. 730-737.
[2]W. Stute, W. G. Manteiga, and M. P. Quindimil (1993). “Bootstrap based goodness-of-fit-tests.” Metrika 40.1: 243-256.
[3]C. Genest, & B Rémillard. (2008). “Validity of the parametric bootstrap for goodness-of-fit testing in semiparametric models.” Annales de l’IHP Probabilités et statistiques. Vol. 44. No. 6.
[4]I. Kojadinovic and J. Yan (2012). “Goodness-of-fit testing based on a weighted bootstrap: A fast large-sample alternative to the parametric bootstrap.” Canadian Journal of Statistics 40.3: 480-500.
[5]B. Phipson and G. K. Smyth (2010). “Permutation P-values Should Never Be Zero: Calculating Exact P-values When Permutations Are Randomly Drawn.” Statistical Applications in Genetics and Molecular Biology 9.1.
[6]H. W. Lilliefors (1967). “On the Kolmogorov-Smirnov test for normality with mean and variance unknown.” Journal of the American statistical Association 62.318: 399-402.
[7]Filliben, James J. “The probability plot correlation coefficient test for normality.” Technometrics 17.1 (1975): 111-117.
範例
一個眾所周知的虛無假設檢定,檢定資料是否來自給定分布,是 Kolmogorov-Smirnov (KS) 檢定,在 SciPy 中以
scipy.stats.ks_1samp提供。假設我們希望檢定以下資料>>> import numpy as np >>> from scipy import stats >>> rng = np.random.default_rng() >>> x = stats.uniform.rvs(size=75, random_state=rng)
是否從常態分布中採樣。為了執行 KS 檢定,觀察到的資料的經驗分布函數將與常態分布的(理論)累積分布函數進行比較。當然,要做到這一點,必須完全指定虛無假設下的常態分布。通常的做法是首先將分布的
loc和scale參數擬合到觀察到的資料,然後執行檢定。>>> loc, scale = np.mean(x), np.std(x, ddof=1) >>> cdf = stats.norm(loc, scale).cdf >>> stats.ks_1samp(x, cdf) KstestResult(statistic=0.1119257570456813, pvalue=0.2827756409939257, statistic_location=0.7751845155861765, statistic_sign=-1)
KS 檢定的一個優點是 p 值 - 在虛無假設下獲得的檢定統計量值與從觀察到的資料獲得的值一樣極端的機率 - 可以精確且有效率地計算出來。
goodness_of_fit只能近似這些結果。>>> known_params = {'loc': loc, 'scale': scale} >>> res = stats.goodness_of_fit(stats.norm, x, known_params=known_params, ... statistic='ks', rng=rng) >>> res.statistic, res.pvalue (0.1119257570456813, 0.2788)
統計量完全匹配,但 p 值是通過形成「蒙地卡羅虛無分布」來估計的,也就是說,通過顯式地從
scipy.stats.norm中抽取隨機樣本並使用提供的參數來計算每個樣本的統計量。這些統計量值中至少與res.statistic一樣極端的比例近似於scipy.stats.ks_1samp計算的精確 p 值。然而,在許多情況下,我們寧願僅檢定資料是否從常態分布族群的任何成員中採樣,而不是專門從位置和尺度擬合到觀察到的樣本的常態分布中採樣。在這種情況下,Lilliefors [6] 認為 KS 檢定過於保守(也就是說,p 值誇大了拒絕真實虛無假設的實際機率),因此缺乏檢定力 - 在虛無假設實際上為假時拒絕虛無假設的能力。實際上,我們上面的 p 值約為 0.28,這對於在任何常見的顯著性水準上拒絕虛無假設來說都太大了。
考慮一下為什麼會這樣。請注意,在上面的 KS 檢定中,統計量始終將資料與擬合到觀察到的資料的常態分布的 CDF 進行比較。這往往會降低觀察到的資料的統計量值,但在計算其他樣本(例如我們隨機抽取以形成蒙地卡羅虛無分布的樣本)的統計量時,這是「不公平的」。很容易糾正這一點:每當我們計算樣本的 KS 統計量時,我們都會使用擬合到該樣本的常態分布的 CDF。在這種情況下,虛無分布尚未精確計算,通常使用如上所述的蒙地卡羅方法進行近似。這正是
goodness_of_fit的優勢所在。>>> res = stats.goodness_of_fit(stats.norm, x, statistic='ks', ... rng=rng) >>> res.statistic, res.pvalue (0.1119257570456813, 0.0196)
確實,這個 p 值要小得多,並且足夠小,可以在常見的顯著性水準(包括 5% 和 2.5%)下(正確地)拒絕虛無假設。
然而,KS 統計量對所有偏離常態性的情況並不是很敏感。KS 統計量的最初優勢是能夠從理論上計算虛無分布,但現在我們可以通過計算方式近似虛無分布,因此可以使用更敏感的統計量 - 從而提高檢定力。Anderson-Darling 統計量 [1] 往往更敏感,並且已使用蒙地卡羅方法針對各種顯著性水準和樣本大小對此統計量的臨界值進行了製表。
>>> res = stats.anderson(x, 'norm') >>> print(res.statistic) 1.2139573337497467 >>> print(res.critical_values) [0.549 0.625 0.75 0.875 1.041] >>> print(res.significance_level) [15. 10. 5. 2.5 1. ]
在這裡,統計量的觀察值超過了與 1% 顯著性水準對應的臨界值。這告訴我們,觀察到的資料的 p 值小於 1%,但它是多少?我們可以從這些(已經內插的)值中內插,但
goodness_of_fit可以直接估計它。>>> res = stats.goodness_of_fit(stats.norm, x, statistic='ad', ... rng=rng) >>> res.statistic, res.pvalue (1.2139573337497467, 0.0034)
另一個優點是
goodness_of_fit的使用不限於一組特定的分布或參數已知與必須從資料估計的條件。相反,對於任何具有足夠快速且可靠的fit方法的分布,goodness_of_fit都可以相對快速地估計 p 值。例如,在這裡,我們使用 Cramer-von Mises 統計量對 Rayleigh 分布執行適合度檢定,其中位置已知,尺度未知。>>> rng = np.random.default_rng() >>> x = stats.chi(df=2.2, loc=0, scale=2).rvs(size=1000, random_state=rng) >>> res = stats.goodness_of_fit(stats.rayleigh, x, statistic='cvm', ... known_params={'loc': 0}, rng=rng)
這執行得相當快,但為了檢查
fit方法的可靠性,我們應該檢查擬合結果。>>> res.fit_result # location is as specified, and scale is reasonable params: FitParams(loc=0.0, scale=2.1026719844231243) success: True message: 'The fit was performed successfully.' >>> import matplotlib.pyplot as plt # matplotlib must be installed to plot >>> res.fit_result.plot() >>> plt.show()
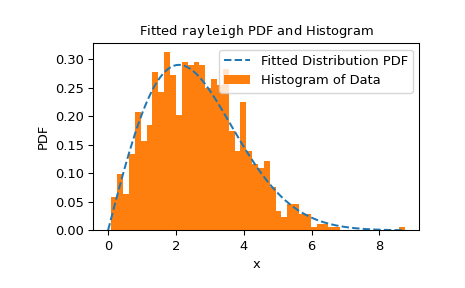
如果分布沒有盡可能好地擬合到觀察到的資料,則檢定可能無法控制第一型錯誤率,也就是說,即使虛無假設為真也拒絕虛無假設的機率。
我們還應該在虛無分布中尋找可能由不可靠的擬合引起的極端離群值。這些不一定會使結果無效,但它們往往會降低檢定的檢定力。
>>> _, ax = plt.subplots() >>> ax.hist(np.log10(res.null_distribution)) >>> ax.set_xlabel("log10 of CVM statistic under the null hypothesis") >>> ax.set_ylabel("Frequency") >>> ax.set_title("Histogram of the Monte Carlo null distribution") >>> plt.show()
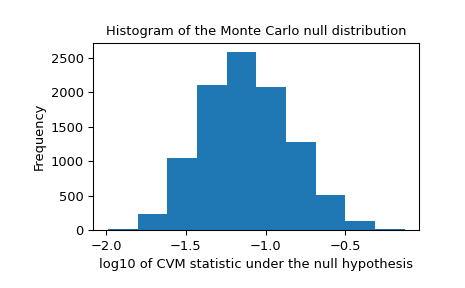
此圖看起來令人放心。
如果
fit方法運作可靠,並且如果檢定統計量的分布對擬合參數的值不是特別敏感,則預計goodness_of_fit提供的 p 值將是一個很好的近似值。>>> res.statistic, res.pvalue (0.2231991510248692, 0.0525)