fit#
- scipy.stats.fit(dist, data, bounds=None, *, guess=None, method='mle', optimizer=<function differential_evolution>)[原始碼]#
將離散或連續分佈擬合到資料
給定一個分佈、資料以及分佈參數的邊界,傳回參數的最大概似估計。
- 參數:
- dist
scipy.stats.rv_continuous或scipy.stats.rv_discrete 代表要擬合到資料的分佈的物件。
- data1D 類陣列
要將分佈擬合到的資料。如果資料包含
np.nan、np.inf或 -np.inf中的任何一個,則 fit 方法將引發ValueError。- bounds字典或元組序列,選用
如果是字典,則每個鍵是分佈的參數名稱,而對應的值是一個元組,其中包含該參數的下限和上限。如果分佈僅針對該參數的有限值範圍定義,則不需要該參數的條目;例如,某些分佈的參數必須在區間 [0, 1] 內。位置 (
loc) 和尺度 (scale) 參數的邊界是選用的;預設情況下,它們分別固定為 0 和 1。如果是序列,則元素 i 是一個元組,其中包含分佈的第 i 個參數的下限和上限。在這種情況下,必須提供所有分佈形狀參數的邊界。或者,位置和尺度的邊界可以跟在分佈形狀參數之後。
如果要固定形狀(例如,如果已知),則下限和上限可以相等。如果使用者提供的下限或上限超出分佈定義域的邊界,則分佈域的邊界將取代使用者提供的值。同樣地,必須是整數的參數將被限制在使用者提供的邊界內的整數值。
- guess字典或類陣列,選用
如果是字典,則每個鍵是分佈的參數名稱,而對應的值是參數值的猜測。
如果是序列,則元素 i 是分佈的第 i 個參數的猜測。在這種情況下,必須提供所有分佈形狀參數的猜測。
如果未提供 guess,則決策變數的猜測不會傳遞給最佳化器。如果提供了 guess,則任何遺失參數的猜測將設定為下限和上限的平均值。必須是整數的參數的猜測將四捨五入為整數值,並且落在使用者提供的邊界和分佈域的交集之外的猜測將被截斷。
- method{‘mle’, ‘mse’}
使用
method="mle"(預設值),擬合是透過最小化負對數概似函數來計算的。對於超出分佈支援範圍的觀測值,會應用較大的有限懲罰(而不是無限負對數概似)。使用method="mse",擬合是透過最小化負對數乘積間隔函數來計算的。對於超出支援範圍的觀測值,會應用相同的懲罰。我們遵循 [1] 的方法,該方法已針對具有重複觀測值的樣本進行了推廣。- optimizer可呼叫物件,選用
optimizer 是一個可呼叫物件,它接受以下位置引數。
- fun可呼叫物件
要最佳化的目標函數。fun 接受一個引數
x,即分佈的候選形狀參數,並傳回給定x、dist 和提供的 data 的目標函數值。optimizer 的工作是找到最小化 fun 的決策變數值。
optimizer 也必須接受以下關鍵字引數。
- bounds元組序列
決策變數值的邊界;每個元素都將是一個元組,其中包含決策變數的下限和上限。
如果提供了 guess,則 optimizer 也必須接受以下關鍵字引數。
- x0類陣列
每個決策變數的猜測。
如果分佈有任何必須是整數的形狀參數,或者如果分佈是離散的且位置參數未固定,則 optimizer 也必須接受以下關鍵字引數。
- integrality布林值類陣列
對於每個決策變數,如果決策變數必須限制為整數值,則為 True,如果決策變數是連續的,則為 False。
optimizer 必須傳回一個物件,例如
scipy.optimize.OptimizeResult的實例,該實例在屬性x中保存決策變數的最佳值。如果提供了屬性fun、status或message,它們將包含在fit傳回的結果物件中。
- dist
- 傳回值:
- result
FitResult 具有以下欄位的物件。
- params具名元組
一個具名元組,其中包含分佈的形狀參數、位置和(如果適用)尺度的最大概似估計。
- success布林值或 None
最佳化器是否認為最佳化已成功終止。
- message字串或 None
最佳化器提供的任何狀態訊息。
該物件具有以下方法
- nllf(params=None, data=None)
預設情況下,給定 data 的擬合 params 處的負對數概似函數。接受包含分佈的替代形狀、位置和尺度的元組,以及替代資料的陣列。
- plot(ax=None)
將擬合分佈的 PDF/PMF 疊加在資料的標準化直方圖上。
- result
另請參閱
註解
當使用者提供包含最大概似估計的嚴格邊界時,最佳化更可能收斂到最大概似估計。例如,當將二項分佈擬合到資料時,可能已知每個樣本的基礎實驗次數,在這種情況下,可以固定對應的形狀參數
n。參考文獻
[1]Shao, Yongzhao, and Marjorie G. Hahn. “Maximum product of spacings method: a unified formulation with illustration of strong consistency.” Illinois Journal of Mathematics 43.3 (1999): 489-499.
範例
假設我們希望將分佈擬合到以下資料。
>>> import numpy as np >>> from scipy import stats >>> rng = np.random.default_rng() >>> dist = stats.nbinom >>> shapes = (5, 0.5) >>> data = dist.rvs(*shapes, size=1000, random_state=rng)
假設我們不知道資料是如何產生的,但我們懷疑它遵循具有參數 n 和 p 的負二項分佈。(請參閱
scipy.stats.nbinom。)我們認為參數 n 小於 30,並且我們知道參數 p 必須位於區間 [0, 1] 內。我們將此資訊記錄在變數 bounds 中,並將此資訊傳遞給fit。>>> bounds = [(0, 30), (0, 1)] >>> res = stats.fit(dist, data, bounds)
fit在使用者指定的 bounds 內搜尋最符合資料的值(以最大概似估計的意義而言)。在這種情況下,它找到的形狀值與資料實際產生的形狀值相似。>>> res.params FitParams(n=5.0, p=0.5028157644634368, loc=0.0) # may vary
我們可以透過將分佈的機率質量函數(具有擬合到資料的形狀)疊加在資料的標準化直方圖上來視覺化結果。
>>> import matplotlib.pyplot as plt # matplotlib must be installed to plot >>> res.plot() >>> plt.show()
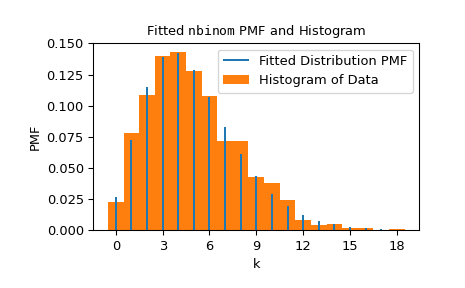
請注意,n 的估計值正好是整數;這是因為
nbinomPMF 的域僅包含整數 n,並且nbinom物件「知道」這一點。nbinom也知道形狀 p 必須是介於 0 和 1 之間的值。在這種情況下 - 當分佈關於參數的域是有限時 - 我們不需要指定參數的邊界。>>> bounds = {'n': (0, 30)} # omit parameter p using a `dict` >>> res2 = stats.fit(dist, data, bounds) >>> res2.params FitParams(n=5.0, p=0.5016492009232932, loc=0.0) # may vary
如果我們希望強制使用固定為 6 的 n 來擬合分佈,我們可以將 n 的下限和上限都設定為 6。但是請注意,在這種情況下,最佳化的目標函數值通常會更差(更高)。
>>> bounds = {'n': (6, 6)} # fix parameter `n` >>> res3 = stats.fit(dist, data, bounds) >>> res3.params FitParams(n=6.0, p=0.5486556076755706, loc=0.0) # may vary >>> res3.nllf() > res.nllf() True # may vary
請注意,先前範例的數值結果是典型的,但它們可能會有所不同,因為
fit使用的預設最佳化器scipy.optimize.differential_evolution是隨機的。但是,我們可以自訂最佳化器使用的設定以確保可重現性 - 甚至完全使用不同的最佳化器 - 使用 optimizer 參數。>>> from scipy.optimize import differential_evolution >>> rng = np.random.default_rng() >>> def optimizer(fun, bounds, *, integrality): ... return differential_evolution(fun, bounds, strategy='best2bin', ... rng=rng, integrality=integrality) >>> bounds = [(0, 30), (0, 1)] >>> res4 = stats.fit(dist, data, bounds, optimizer=optimizer) >>> res4.params FitParams(n=5.0, p=0.5015183149259951, loc=0.0)