linkage#
- scipy.cluster.hierarchy.linkage(y, method='single', metric='euclidean', optimal_ordering=False)[原始碼]#
執行階層式/聚合式分群。
輸入 y 可以是 1-D 壓縮距離矩陣或 2-D 觀測向量陣列。
如果 y 是 1-D 壓縮距離矩陣,則 y 必須是 \(\binom{n}{2}\) 大小的向量,其中 n 是距離矩陣中配對的原始觀測數量。此函數的行為與 MATLAB linkage 函數非常相似。
返回一個 \((n-1)\) 乘 4 的矩陣
Z。在第 \(i\) 次迭代中,索引為Z[i, 0]和Z[i, 1]的群集被合併以形成群集 \(n + i\)。索引小於 \(n\) 的群集對應於 \(n\) 個原始觀測值之一。群集Z[i, 0]和Z[i, 1]之間的距離由Z[i, 2]給出。第四個值Z[i, 3]表示新形成的群集中原始觀測值的數量。以下連結方法用於計算兩個群集 \(s\) 和 \(t\) 之間的距離 \(d(s, t)\)。該演算法從尚未在形成的層次結構中使用的群集森林開始。當森林中的兩個群集 \(s\) 和 \(t\) 組合為單個群集 \(u\) 時,\(s\) 和 \(t\) 從森林中移除,並且 \(u\) 被添加到森林中。當森林中僅剩一個群集時,演算法停止,並且該群集成為根。
在每次迭代中維護一個距離矩陣。
d[i,j]條目對應於原始森林中群集 \(i\) 和 \(j\) 之間的距離。在每次迭代中,演算法必須更新距離矩陣,以反映新形成的群集 u 與森林中剩餘群集的距離。
假設群集 \(u\) 中有 \(|u|\) 個原始觀測值 \(u[0], \ldots, u[|u|-1]\),並且群集 \(v\) 中有 \(|v|\) 個原始物件 \(v[0], \ldots, v[|v|-1]\)。回想一下,\(s\) 和 \(t\) 組合形成群集 \(u\)。令 \(v\) 為森林中不是 \(u\) 的任何剩餘群集。
以下是計算新形成的群集 \(u\) 與每個 \(v\) 之間距離的方法。
method=’single’ 指定
\[d(u,v) = \min(dist(u[i],v[j]))\]對於群集 \(u\) 中的所有點 \(i\) 和群集 \(v\) 中的 \(j\)。這也稱為最近點演算法。
method=’complete’ 指定
\[d(u, v) = \max(dist(u[i],v[j]))\]對於群集 u 中的所有點 \(i\) 和群集 \(v\) 中的 \(j\)。這也稱為最遠點演算法或 Voor Hees 演算法。
method=’average’ 指定
\[d(u,v) = \sum_{ij} \frac{d(u[i], v[j])} {(|u|*|v|)}\]對於所有點 \(i\) 和 \(j\),其中 \(|u|\) 和 \(|v|\) 分別是群集 \(u\) 和 \(v\) 的基數。這也稱為 UPGMA 演算法。
method=’weighted’ 指定
\[d(u,v) = (dist(s,v) + dist(t,v))/2\]其中群集 u 是由群集 s 和 t 形成的,而 v 是森林中的剩餘群集(也稱為 WPGMA)。
method=’centroid’ 指定
\[dist(s,t) = ||c_s-c_t||_2\]其中 \(c_s\) 和 \(c_t\) 分別是群集 \(s\) 和 \(t\) 的質心。當兩個群集 \(s\) 和 \(t\) 組合為一個新群集 \(u\) 時,新的質心是根據群集 \(s\) 和 \(t\) 中的所有原始物件計算的。然後,距離變成 \(u\) 的質心與森林中剩餘群集 \(v\) 的質心之間的歐幾里得距離。這也稱為 UPGMC 演算法。
method=’median’ 指定 \(d(s,t)\) 類似於
centroid方法。當兩個群集 \(s\) 和 \(t\) 組合為一個新群集 \(u\) 時,質心 s 和 t 的平均值給出新的質心 \(u\)。這也稱為 WPGMC 演算法。method=’ward’ 使用 Ward 變異數最小化演算法。新條目 \(d(u,v)\) 計算如下:
\[d(u,v) = \sqrt{\frac{|v|+|s|} {T}d(v,s)^2 + \frac{|v|+|t|} {T}d(v,t)^2 - \frac{|v|} {T}d(s,t)^2}\]其中 \(u\) 是由群集 \(s\) 和 \(t\) 組成的新合併群集,\(v\) 是森林中未使用的群集,\(T=|v|+|s|+|t|\),而 \(|*|\) 是其引數的基數。這也稱為增量演算法。
警告:當選擇森林中的最小距離對時,可能存在兩個或更多對具有相同的最小距離。此實作可能會選擇與 MATLAB 版本不同的最小值。
- 參數:
- yndarray
壓縮距離矩陣。壓縮距離矩陣是一個平面陣列,包含距離矩陣的上三角部分。這是
pdist返回的形式。或者,可以將 \(n\) 維中的 \(m\) 個觀測向量的集合作為 \(m\) 乘 \(n\) 陣列傳遞。壓縮距離矩陣的所有元素都必須是有限的,即,沒有 NaN 或 inf。- methodstr,可選
要使用的連結演算法。有關完整說明,請參閱下面的
連結方法部分。- metricstr 或函數,可選
當 y 是觀測向量集合時要使用的距離度量;否則忽略。有關有效距離度量的列表,請參閱
pdist函數。也可以使用自訂距離函數。- optimal_orderingbool,可選
如果為 True,則將重新排序連結矩陣,以使連續葉子之間的距離最小。當資料視覺化時,這會產生更直觀的樹狀結構。預設為 False,因為此演算法可能很慢,尤其是在大型資料集上 [2]。另請參閱
optimal_leaf_ordering函數。在 1.0.0 版本中新增。
- 返回:
- Zndarray
階層式分群編碼為連結矩陣。
參見
筆記
對於 ‘single’ 方法,實作了基於最小生成樹的優化演算法。它的時間複雜度為 \(O(n^2)\)。對於 ‘complete’、‘average’、‘weighted’ 和 ‘ward’ 方法,實作了稱為最近鄰鏈的演算法。它的時間複雜度也為 \(O(n^2)\)。對於其他方法,實作了時間複雜度為 \(O(n^3)\) 的樸素演算法。所有演算法都使用 \(O(n^2)\) 記憶體。有關演算法的詳細資訊,請參閱 [1]。
僅當使用歐幾里得成對度量時,才能正確定義 ‘centroid’、‘median’ 和 ‘ward’ 方法。如果 y 作為預先計算的成對距離傳遞,則使用者有責任確保這些距離實際上是歐幾里得距離,否則產生的結果將不正確。
參考文獻
[1]Daniel Mullner,“現代階層式聚合分群演算法”,arXiv:1109.2378v1。
[2]Ziv Bar-Joseph、David K. Gifford、Tommi S. Jaakkola,“階層式分群的快速最佳葉排序”,2001 年。生物資訊學 DOI:10.1093/bioinformatics/17.suppl_1.S22
範例
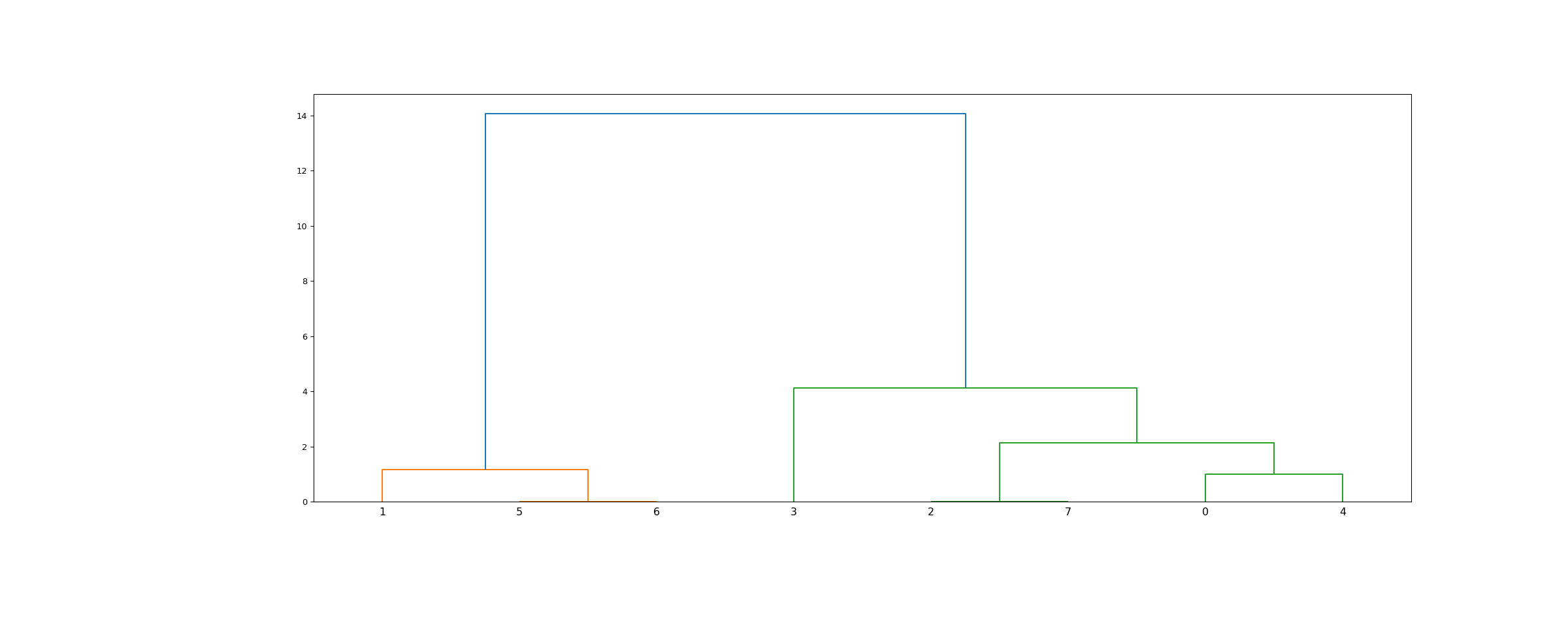
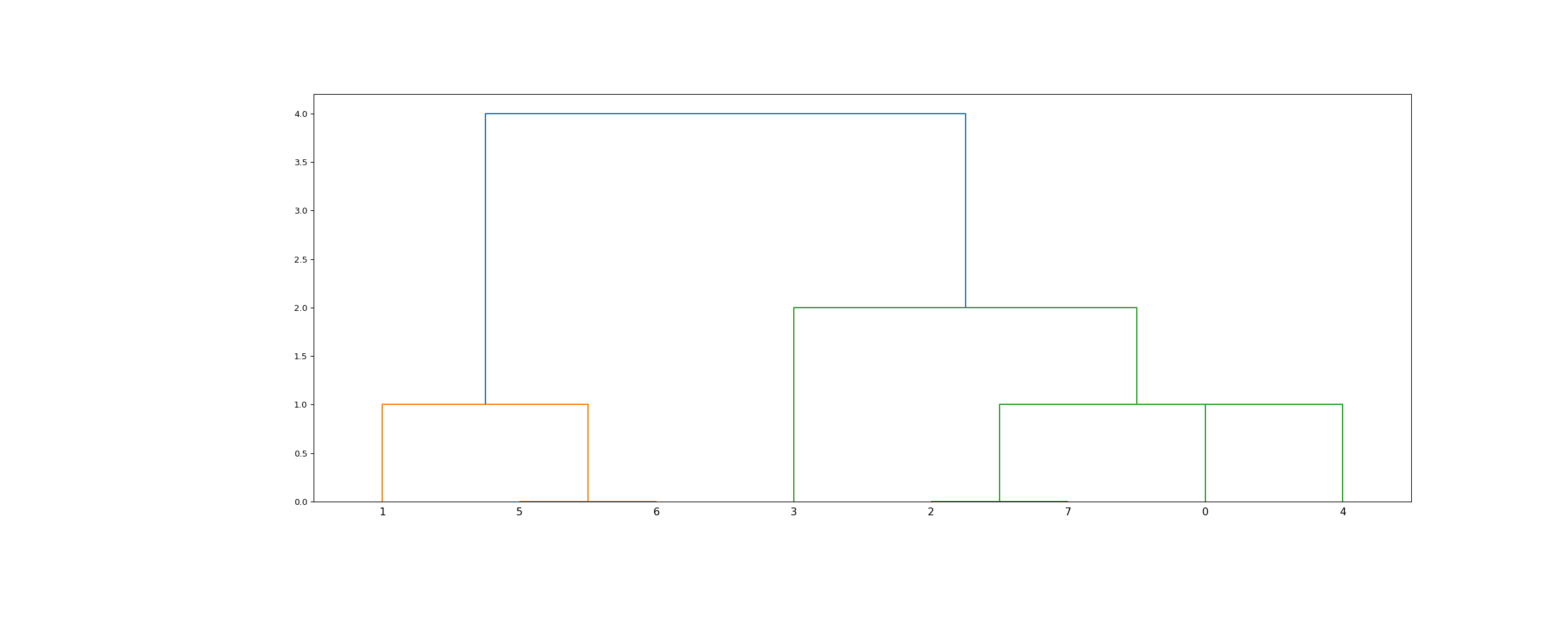
>>> from scipy.cluster.hierarchy import dendrogram, linkage >>> from matplotlib import pyplot as plt >>> X = [[i] for i in [2, 8, 0, 4, 1, 9, 9, 0]]
>>> Z = linkage(X, 'ward') >>> fig = plt.figure(figsize=(25, 10)) >>> dn = dendrogram(Z)
>>> Z = linkage(X, 'single') >>> fig = plt.figure(figsize=(25, 10)) >>> dn = dendrogram(Z) >>> plt.show()