隨機變數轉換指南#
背景#
在 SciPy 1.15 之前,SciPy 的所有連續機率分佈(例如 scipy.stats.norm)都是 scipy.stats.rv_continuous 子類別的實例。
from scipy import stats
dist = stats.norm
type(dist)
scipy.stats._continuous_distns.norm_gen
isinstance(dist, stats.rv_continuous)
True
有兩種明顯的方式來處理這些物件。
根據更常見的方式,將「引數」(例如 x)和「分佈參數」(例如 loc、scale)作為物件方法的輸入提供。
x, loc, scale = 1., 0., 1.
dist.pdf(x, loc, scale)
0.24197072451914337
較不常見的方法是調用分佈物件的 __call__ 方法,無論原始類別為何,該方法都會傳回 rv_continuous_frozen 的實例。
frozen = stats.norm()
type(frozen)
scipy.stats._distn_infrastructure.rv_continuous_frozen
此新物件的方法僅接受引數,而不接受分佈參數。
frozen.pdf(x)
0.24197072451914337
從某種意義上說,rv_continuous 的實例(如 norm)代表「分佈族」,需要參數來識別特定的機率分佈,而 rv_continuous_frozen 的實例則類似於「隨機變數」——一個遵循特定機率分佈的數學物件。
這兩種方法都是有效的,並且在某些情況下具有優勢。例如,對於如此簡單的調用或當方法被視為分佈參數的函數而不是通常的引數(例如,似然性)時,stats.norm.pdf(x) 看起來比 stats.norm().pdf(x) 更自然。然而,前一種方法有一些固有的缺點;例如,SciPy 的所有 125 個連續分佈都必須在匯入時實例化,每次調用方法時都必須驗證分佈參數,並且方法的文檔必須 a) 為每個分佈的每個方法單獨生成,或 b) 省略每個分佈獨有的形狀參數。為了解決這些和其他缺點,gh-15928 提出了一種基於後者(隨機變數)方法的新型獨立基礎架構。本轉換指南記錄了 rv_continuous 和 rv_continuous_frozen 的使用者如何遷移到新的基礎架構。
注意:對於某些用例,尤其是將分佈參數擬合到數據,新的基礎架構可能還不夠方便。如果舊的基礎架構符合使用者的需求,歡迎繼續使用;目前尚未棄用。
基本概念#
在新的基礎架構中,分佈族是根據 CamelCase 慣例命名的類別。它們必須在使用前實例化,參數以僅限關鍵字引數傳遞。分佈族類別的實例可以被認為是隨機變數,在數學中通常使用大寫字母表示。
from scipy import stats
mu, sigma = 0, 1
X = stats.Normal(mu=mu, sigma=sigma)
X
Normal(mu=0.0, sigma=1.0)
實例化後,形狀參數可以作為屬性讀取(但不能寫入)。
X.mu, X.sigma
(0.0, 1.0)
scipy.stats.Normal 的文檔包含指向其每個方法的詳細文檔的連結。(例如,與 scipy.stats.norm 的文檔進行比較。)
注意:儘管截至 SciPy 1.15,只有 Normal 和 Uniform 具有渲染的文檔,並且可以直接從 stats 匯入,但幾乎所有其他舊分佈都可以透過 make_distribution(稍後討論)與新介面一起使用。
然後,透過僅傳遞引數(如果有的話)而不是分佈參數來調用方法。
X.mean()
0.0
X.pdf(x)
0.24197072451914337
對於像這樣簡單的調用(例如,引數是有效的浮點數),調用新隨機變數的方法通常比調用舊分佈方法更快。
%timeit X.pdf(x)
2.47 µs ± 5.18 ns per loop (mean ± std. dev. of 7 runs, 100,000 loops each)
%timeit frozen.pdf(x)
21.8 µs ± 161 ns per loop (mean ± std. dev. of 7 runs, 10,000 loops each)
%timeit dist.pdf(x, loc=mu, scale=sigma)
22 µs ± 21.2 ns per loop (mean ± std. dev. of 7 runs, 10,000 loops each)
請注意,X.pdf 和 frozen.pdf 的調用方式是相同的,而 dist.pdf 的調用方式非常相似——唯一的區別在於對 dist.pdf 的調用包含形狀參數,而在新的基礎架構中,形狀參數僅在實例化隨機變數時提供。
除了 pdf 和 mean 之外,新基礎架構的其他幾種方法與舊方法基本相同。
logpdf(機率密度函數的對數)cdf(累積分佈函數)logcdf(累積分佈函數的對數)entropy(微分熵)中位數支持
其他方法有新的名稱,但否則是可以直接替換的。
sf(生存函數) \(\rightarrow\)ccdf(互補累積分佈函數)logsf\(\rightarrow\)logccdfppf(百分點函數) \(\rightarrow\)icdf(反向累積分佈函數)isf(反向生存函數) \(\rightarrow\)iccdf(反向互補累積分佈函數)std\(\rightarrow\)standard_deviation(標準差)var\(\rightarrow\)variance(變異數)
新的基礎架構有幾個與上述相同方向的新方法。
ilogcdf(累積分佈函數對數的反函數)ilogccdf(互補累積分佈函數對數的反函數)logentropy(熵的對數)mode(分佈的眾數)偏度kurtosis(非超額峰度;請參閱下方的「標準化動差」)
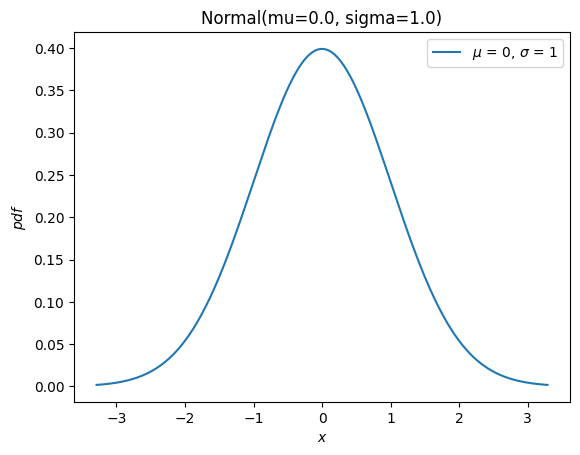
它還有一個方便的新 plot 方法
import matplotlib.pyplot as plt
X.plot()
plt.show()
舊基礎架構的大部分剩餘方法(rvs、moment、stats、interval、fit、nnlf、fit_loc_scale 和 expect)具有類似的功能,但需要謹慎處理。在描述替換方法之前,我們先簡要介紹如何使用非常態分佈的隨機變數:幾乎所有舊的分佈物件都可以使用 scipy.stats.make_distribution 轉換為新的分佈類別,並且可以透過將形狀參數作為關鍵字引數傳遞來實例化新的分佈類別。例如,考慮 Weibull 分佈。我們可以建立一個新的類別,它是分佈族的抽象,如下所示
Weibull = stats.make_distribution(stats.weibull_min)
print(Weibull.__doc__[:288]) # help(Weibull) prints this and (too much) more
This class represents `scipy.stats.weibull_min` as a subclass of `ContinuousDistribution`.
The `repr`/`str` of class instances is `Weibull`.
The PDF of the distribution is defined for :math:`x` in [0.0, \infty].
This class accepts one parameterization:
`c` for :math:`c \in (0, \infty)`.
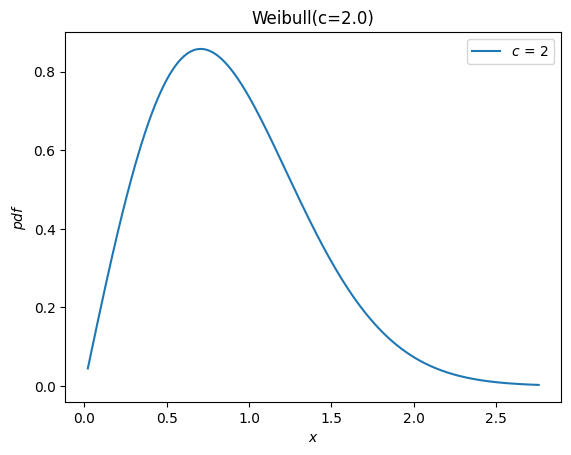
根據 weibull_min 的文檔,形狀參數表示為 c,因此我們可以透過將 c 作為關鍵字引數傳遞給新的 Weibull 類別來實例化隨機變數。
c = 2.
X = Weibull(c=c)
X.plot()
plt.show()
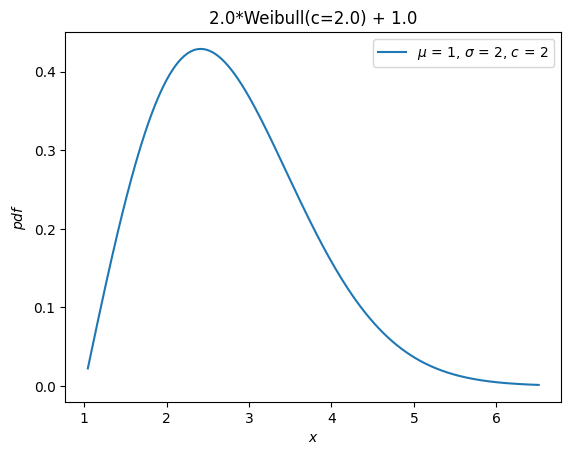
以前,所有分佈都繼承了 loc 和 scale 分佈參數。這些不再被接受。相反,隨機變數可以使用算術運算符進行平移和縮放。
Y = 2*X + 1
Y.plot() # note the change in the abscissae
plt.show()
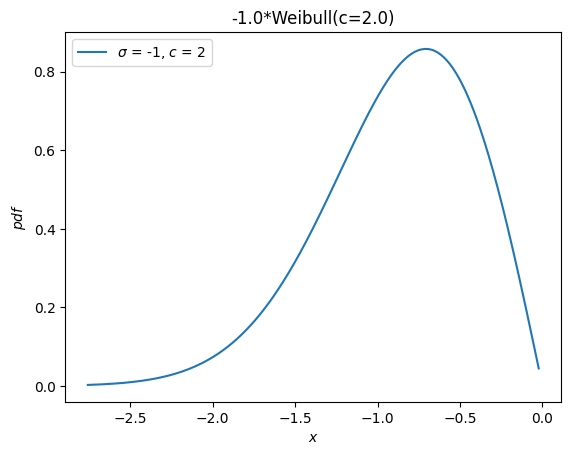
一個獨立的分佈 weibull_max 作為 weibull_min 關於原點的反射而提供。現在,這只是 -X。
Y = -X
Y.plot()
plt.show()
動差#
舊的基礎架構為原始動差提供了一個 moments 方法。當僅指定階數時,新的 moment 方法是直接替換。
stats.weibull_min.moment(1, c), X.moment(1) # first raw moment of the Weibull distribution with shape c
(0.8862269254527579, 0.8862269254527579)
但是,舊的基礎架構也有一個 stats 方法,它提供了分佈的各種統計量。以下統計量與指示的字元相關聯。
平均值(關於原點的第一個原始動差,
'm')變異數(第二個中心動差,
'v')偏度(第三個標準化動差,
's')超額峰度(第四個標準化動差減 3,
'k')
例如
stats.weibull_min.stats(c, moments='mvsk')
(0.8862269254527579,
0.21460183660255183,
0.6311106578189344,
0.24508930068764556)
現在,可以透過將適當的引數傳遞給新的 moment 方法來計算任何 order 和 kind(原始、中心和標準化)的動差。
(X.moment(order=1, kind='raw'),
X.moment(order=2, kind='central'),
X.moment(order=3, kind='standardized'),
X.moment(order=4, kind='standardized'))
(0.8862269254527579,
0.21460183660255183,
0.6311106578189344,
3.2450893006876314)
請注意 峰度 定義的差異。以前,提供了「超額」(又稱「Fisher's」)峰度。作為一種慣例(而不是自然的數學定義),這是透過常數值 (3) 平移的標準化第四動差,以給出常態分佈的 0.0 值。
stats.norm.stats(moments='k')
0.0
新的 moment 和 kurtosis 方法預設不遵守此慣例;它們根據數學定義報告標準化第四動差。這也稱為「非超額」(或「Pearson's」)峰度,對於標準常態分佈,其值為 3.0。
stats.Normal().moment(4, kind='standardized')
3.0
為了方便起見,kurtosis 方法提供了一個 convention 引數,用於在兩者之間進行選擇。
stats.Normal().kurtosis(convention='excess'), stats.Normal().kurtosis(convention='non-excess')
(0.0, 3.0)
隨機變量#
舊基礎架構的 rvs 方法已替換為 sample,但是當 1) 需要控制隨機狀態或 2) 將陣列用於形狀、位置或縮放參數時,應注意兩個差異。
首先,根據 SPEC 7,引數 random_state 已替換為 rng。過去控制隨機狀態的一種模式是使用 numpy.random.seed 或將整數種子傳遞給 random_state 引數。整數種子被轉換為 numpy.random.RandomState 的實例,因此在兩種情況下,給定整數種子的行為將是相同的
import numpy as np
np.random.seed(1)
dist = stats.norm
dist.rvs(), dist.rvs(random_state=1)
(1.6243453636632417, 1.6243453636632417)
在新基礎架構中,將 numpy.Generator 的實例傳遞給 sample 的 rng 引數以控制隨機狀態。請注意,整數種子現在被轉換為 numpy.random.Generator 的實例,而不是 numpy.random.RandomState 的實例。
X = stats.Normal()
rng = np.random.default_rng(1) # instantiate a numpy.random.Generator
X.sample(rng=rng), X.sample(rng=1)
(0.345584192064786, 0.345584192064786)
其次,引數 shape 替換了引數 size。
dist.rvs(size=(3, 4))
array([[-0.61175641, -0.52817175, -1.07296862, 0.86540763],
[-2.3015387 , 1.74481176, -0.7612069 , 0.3190391 ],
[-0.24937038, 1.46210794, -2.06014071, -0.3224172 ]])
X.sample(shape=(3, 4))
array([[ 1.09030882, 1.00534614, 2.31221283, -1.19646394],
[ 0.68798893, 0.75769842, 1.52220417, 0.3066763 ],
[ 1.15948958, 1.51430283, -1.01990881, -0.14666494]])
除了名稱上的差異外,當涉及陣列形狀參數時,行為也存在差異。以前,分佈參數陣列的形狀必須包含在 size 中。
dist.rvs(size=(3, 4, 2), loc=[0, 1]).shape # `loc` has shape (2,)
(3, 4, 2)
現在,參數陣列的形狀被認為是隨機變數物件本身的屬性。指定陣列形狀參數的形狀將是多餘的,因此在指定樣本的 shape 時不包含它。
Y = stats.Normal(mu = [0, 1])
Y.sample(shape=(3, 4)).shape # the sample has shape (3, 4); each element is of shape (2,)
(3, 4, 2)
機率質量(又稱「信賴」)區間#
舊的基礎架構提供了一個 interval 方法,預設情況下,該方法將傳回一個對稱(關於中位數)區間,其中包含指定百分比的分佈機率質量。
a = 0.95
dist.interval(confidence=a)
(-1.959963984540054, 1.959963984540054)
現在,使用每個尾部所需的機率質量調用反向 CDF 和互補 CDF 方法。
p = 1 - a
X.icdf(p/2), X.iccdf(p/2)
(-1.959963984540054, 1.959963984540054)
似然函數#
舊的基礎架構提供了一個函數來計算負對數似然函數,錯誤地命名為 nnlf(而不是 nllf)。它接受分佈的參數作為元組,並接受觀測值作為陣列。
mu = 0
sigma = 1
data = stats.norm.rvs(size=100, loc=mu, scale=sigma)
stats.norm.nnlf((mu, sigma), data)
131.07116392314364
現在,只需根據其數學定義計算負對數似然性。
X = stats.Normal(mu=mu, sigma=sigma)
-X.logpdf(data).sum()
131.07116392314364
期望值#
舊基礎架構的 expect 方法估計任意函數的定積分,該函數由分佈的 PDF 加權。例如,分佈的第四動差由下式給出
def f(x): return x**4
stats.norm.expect(f, lb=-np.inf, ub=np.inf)
3.000000000000001
與原始碼執行的操作相比,這幾乎沒有增加便利性:使用 scipy.integrate.quad 執行數值積分。
from scipy import integrate
def f(x): return x**4 * stats.norm.pdf(x)
integrate.quad(f, a=-np.inf, b=np.inf) # integral estimate, estimate of the error
(3.0000000000000053, 1.2043244026618655e-08)
當然,這也可以很容易地使用新的基礎架構來完成。
def f(x): return x**4 * X.pdf(x)
integrate.quad(f, a=-np.inf, b=np.inf) # integral estimate, estimate of the error
(3.0000000000000053, 1.2043244043338238e-08)
conditional 引數只是將結果按區間內包含的機率質量的倒數進行縮放。
a, b = -1, 3
def f(x): return x**4
stats.norm.expect(f, lb=a, ub=b, conditional=True)
1.657813862143119
直接使用 quad,即
def f(x): return x**4 * stats.norm.pdf(x)
prob = stats.norm.cdf(b) - stats.norm.cdf(a)
integrate.quad(f, a=a, b=b)[0] / prob
1.6578138621431193
使用新的隨機變數
integrate.quad(f, a=a, b=b)[0] / X.cdf(a, b)
1.657813862143119
請注意,新的隨機變數的 cdf 方法接受兩個引數來計算指定區間內的機率質量。在許多情況下,這可以避免當機率質量非常小時的減法誤差(「災難性抵消」)。
擬合#
位置/縮放估計#
舊的基礎架構提供了一個函數,用於從數據中估計分佈的位置和縮放參數。
rng = np.random.default_rng(91392794601852341588152500276639671056)
dist = stats.weibull_min
c, loc, scale = 0.5, 0, 4.
data = dist.rvs(size=100, c=c, loc=loc, scale=scale, random_state=rng)
dist.fit_loc_scale(data, c)
# compare against 0 (default location) and 4 (specified scale)
(1.8268432669973347, 1.9640905288934327)
根據原始碼,很容易複製該方法。
X = Weibull(c=c)
def fit_loc_scale(X, data):
m, v = X.mean(), X.variance()
m_, v_ = data.mean(), data.var()
scale = np.sqrt(v_ / v)
loc = m_ - scale*m
return loc, scale
fit_loc_scale(X, data)
(1.8268432669973347, 1.9640905288934327)
請注意,在此範例中,估計值非常差,並且啟發式方法的效能不佳已納入不提供替換方法的決策中。
最大似然估計#
舊基礎架構的最後一個方法是 fit,它在給定分佈樣本的情況下估計基礎分佈的位置、縮放和形狀參數。
c_, loc_, scale_ = stats.weibull_min.fit(data)
c_, loc_, scale_
(0.5960699537061247, 1.8950104942593064e-05, 4.015875612107546)
此方法的便利性既是祝福也是詛咒。
當它工作時,簡單性非常受歡迎。對於某些分佈,已手動編寫參數的 最大似然估計 (MLE) 的解析表達式,對於這些分佈,fit 方法非常可靠。
但是,對於大多數分佈,擬合是透過負對數似然函數的數值最小化來執行的。這不能保證成功——既因為 MLE 的固有局限性,也因為數值最佳化的最新技術——在這些情況下,fit 的使用者會陷入困境。但是,對其原理的少量理解對於確保成功大有幫助,因此在這裡我們介紹一個使用新基礎架構進行擬合的迷你教程。
首先,請注意 MLE 是將分佈擬合到數據的幾種潛在策略之一。它所做的只是找到使負對數似然性 (NLLF) 最小化的分佈參數值。請注意,對於從中生成數據的分佈,NLLF 為
stats.weibull_min.nnlf((c, loc, scale), data)
246.9503871545133
使用 fit 估計的參數的 NLLF 較低
stats.weibull_min.nnlf((c_, loc_, scale_), data)
230.35385152369787
因此,無論這是否滿足使用者對「良好擬合」的概念,或者參數是否在合理的範圍內,fit 都認為它的工作已完成。除了允許使用者指定分佈參數的猜測外,它對過程的控制很少。(注意:從技術上講,optimizer 引數可用於更大的控制,但這沒有很好的文檔記錄,並且其使用抵消了 fit 方法提供的任何便利性。)
另一個問題是,對於某些分佈,MLE 本質上表現不佳。例如,在此範例中,我們可以透過簡單地將位置設定為數據的最小值來使 NLLF 盡可能低——即使對於形狀和縮放參數的虛假值也是如此。
stats.weibull_min.nnlf((0.1, np.min(data), 10), data)
-inf
與這些問題複合的是,fit 預設情況下估計所有分佈參數,包括位置。在上面的範例中,我們可能只對更常見的雙參數 Weibull 分佈感興趣,因為實際位置為零。fit 可以透過指定位置是固定參數來適應這一點。
# c_, loc_, scale_ = stats.weibull_min.fit(data, loc=0) # careful! this provides loc=0 as a *guess*
c_, loc_, scale_ = stats.weibull_min.fit(data, floc=0)
c_, loc_, scale_
(0.5834128280886433, 0, 3.829972848420729)
stats.weibull_min.nnlf((c_, loc_, scale_), data)
244.9617429249505
但是,透過提供據稱「只需工作」的 fit 方法,鼓勵使用者忽略其中一些重要細節。
相反,我們建議使用 SciPy 提供的通用最佳化工具將分佈擬合到數據幾乎同樣簡單。但是,這允許使用者根據其首選目標函數執行擬合,在事情未按預期工作時提供選項,並幫助使用者避免最常見的陷阱。
例如,假設我們期望的目標函數確實是負對數似然性。將其定義為僅形狀參數 c 和縮放比例的函數非常簡單,將位置保留為預設值零。
def nllf(x):
c, scale = x # pass (only) `c` and `scale` as elements of `x`
X = Weibull(c=c) * scale
return -X.logpdf(data).sum()
nllf((c_, scale_))
244.96174292495053
為了執行最小化,我們可以使用 scipy.optimize.minimize。
from scipy import optimize
eps = 1e-10 # numerical tolerance to avoid invalid parameters
lb = [eps, eps] # lower bound on `c` and `scale`
ub = [10, 10] # upper bound on `c` and `scale`
x0 = [1, 1] # guess to get optimization started
bounds = optimize.Bounds(lb, ub)
res_mle = optimize.minimize(nllf, x0, bounds=bounds)
res_mle
message: CONVERGENCE: NORM OF PROJECTED GRADIENT <= PGTOL
success: True
status: 0
fun: 244.96174292277675
x: [ 5.834e-01 3.830e+00]
nit: 13
jac: [ 5.684e-06 0.000e+00]
nfev: 45
njev: 15
hess_inv: <2x2 LbfgsInvHessProduct with dtype=float64>
目標函數的值基本相同,並且 PDF 無法區分。
import matplotlib.pyplot as plt
x = np.linspace(eps, 15, 300)
c_, scale_ = res_mle.x
X = Weibull(c=c_)*scale_
plt.plot(x, X.pdf(x), '-', label='numerical optimization')
c_, _, scale_ = stats.weibull_min.fit(data, floc=0)
Y = Weibull(c=c_)*scale_
plt.plot(x, Y.pdf(x), '--', label='`weibull_min.fit`')
plt.hist(data, bins=np.linspace(0, 20, 30), density=True, alpha=0.1)
plt.xlabel('x')
plt.ylabel('pdf(x)')
plt.legend()
plt.ylim(0, 0.5)
plt.show()
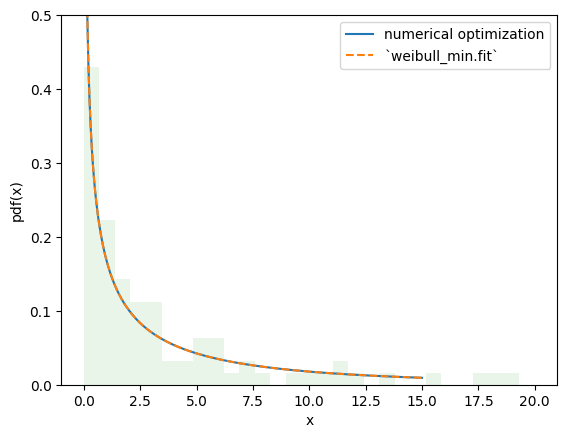
但是,使用這種方法,可以輕鬆地對擬合程序進行更改以滿足我們的需求,從而實現 rv_continuous.fit 提供的擬合程序以外的其他估計程序。例如,我們可以透過將目標函數更改為負對數乘積間距來執行 最大間距估計 (MSE),如下所示。
def nlps(x):
c, scale = x
X = Weibull(c=c) * scale
x = np.sort(np.concatenate((data, X.support()))) # Append the endpoints of the support to the data
return -X.logcdf(x[:-1], x[1:]).sum().real # Minimize the sum of the logs the probability mass between points
res_mps = optimize.minimize(nlps, x0, bounds=bounds)
res_mps.x
array([0.55917917, 3.85552195])
對於 L-動差 方法(嘗試匹配分佈和樣本 L-動差)
def lmoment_residual(x):
c, scale = x
X = Weibull(c=c) * scale
E11 = stats.order_statistic(X, r=1, n=1).mean()
E12, E22 = stats.order_statistic(X, r=[1, 2], n=2).mean()
lmoments_X = [E11, 0.5*(E22 - E12)] # the first two l-moments of the distribution
lmoments_x = stats.lmoment(data, order=[1, 2]) # first two l-moments of the data
return np.linalg.norm(lmoments_x - lmoments_X) # Minimize the norm of the difference
x0 = [0.4, 3] # This method is a bit sensitive to the initial guess
res_lmom = optimize.minimize(lmoment_residual, x0, bounds=bounds)
res_lmom.x, res_lmom.fun # residual should be ~0
(array([0.60943275, 3.90234501]), 5.058079590921588e-07)
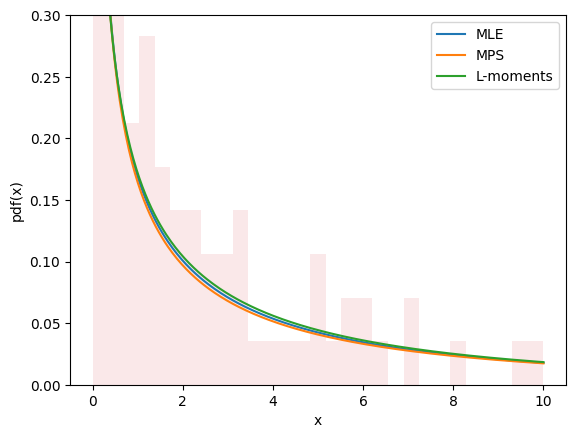
所有三種方法的圖看起來都很相似,並且似乎描述了數據,這讓我們對我們有一個良好的擬合有一定信心。
import matplotlib.pyplot as plt
x = np.linspace(eps, 10, 300)
for res, label in [(res_mle, "MLE"), (res_mps, "MPS"), (res_lmom, "L-moments")]:
c_, scale_ = res.x
X = Weibull(c=c_)*scale_
plt.plot(x, X.pdf(x), '-', label=label)
plt.hist(data, bins=np.linspace(0, 10, 30), density=True, alpha=0.1)
plt.xlabel('x')
plt.ylabel('pdf(x)')
plt.legend()
plt.ylim(0, 0.3)
plt.show()
估計並不總是涉及將分佈擬合到數據。例如,在 gh-12134 中,使用者需要計算 Weibull 分佈的形狀和縮放參數以達到給定的平均值和標準差。這很容易融入相同的框架。
moments_0 = np.asarray([8, 20]) # desired mean and standard deviation
def f(x):
c, scale = x
X = Weibull(c=c) * scale
moments_X = X.mean(), X.standard_deviation()
return np.linalg.norm(moments_X - moments_0)
bounds.lb = np.asarray([0.1, 0.1]) # the Weibull distribution is problematic for very small c
res = optimize.minimize(f, x0, bounds=bounds, method='slsqp') # easily change the minimization method
res
message: Optimization terminated successfully
success: True
status: 0
fun: 4.595040620463497e-07
x: [ 4.627e-01 3.430e+00]
nit: 21
jac: [ 1.322e+02 -2.328e-01]
nfev: 100
njev: 21
新的和進階功能#
轉換#
數學轉換可以應用於隨機變數。例如,許多基本算術運算(+、-、*、/、**)在隨機變數和純量之間起作用。
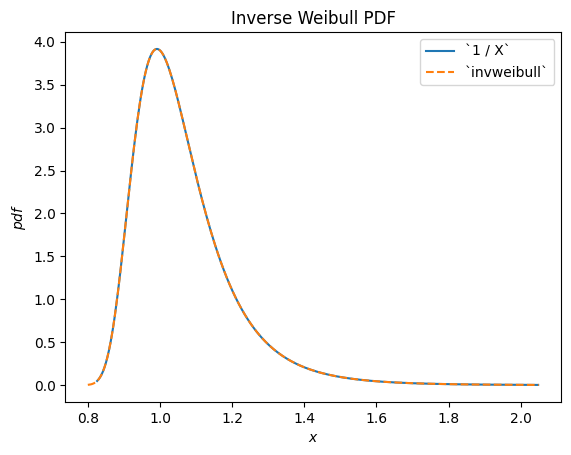
例如,我們可以看到 Weibull 分佈的 RV 的倒數根據 scipy.stats.invweibull 分佈。(「反向」分佈通常是隨機變數倒數的基礎分佈。)
c = 10.6
X = Weibull(c=10.6)
Y = 1 / X # compare to `invweibull`
Y.plot()
x = np.linspace(0.8, 2.05, 300)
plt.plot(x, stats.invweibull(c=c).pdf(x), '--')
plt.legend(['`1 / X`', '`invweibull`'])
plt.title("Inverse Weibull PDF")
plt.show()
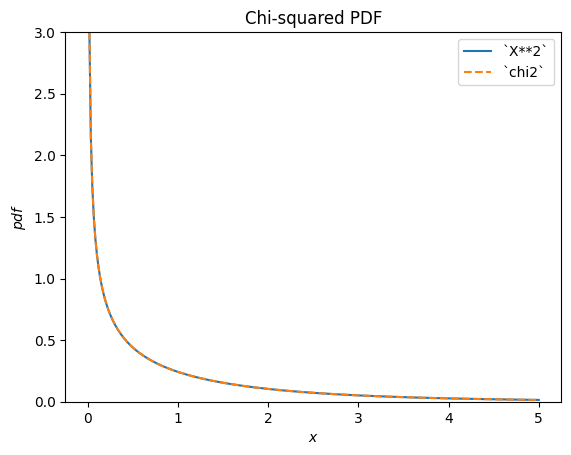
scipy.stats.chi2 描述了常態分佈隨機變數的平方和。
X = stats.Normal()
Y = X**2 # compare to chi2
Y.plot(t=('x', eps, 5));
x = np.linspace(eps, 5, 300)
plt.plot(x, stats.chi2(df=1).pdf(x), '--')
plt.ylim(0, 3)
plt.legend(['`X**2`', '`chi2`'])
plt.title("Chi-squared PDF")
plt.show()
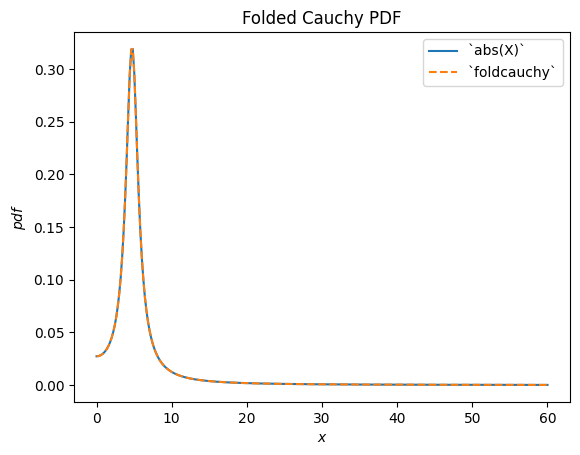
scipy.stats.foldcauchy 描述了 Cauchy 分佈隨機變數的絕對值。(「摺疊」分佈是隨機變數絕對值的基礎分佈。)
Cauchy = stats.make_distribution(stats.cauchy)
c = 4.72
X = Cauchy() + c
Y = abs(X) # compare to `foldcauchy`
Y.plot(t=('x', 0, 60))
x = np.linspace(0, 60, 300)
plt.plot(x, stats.foldcauchy(c=c).pdf(x), '--')
plt.legend(['`abs(X)`', '`foldcauchy`'])
plt.title("Folded Cauchy PDF")
plt.show()
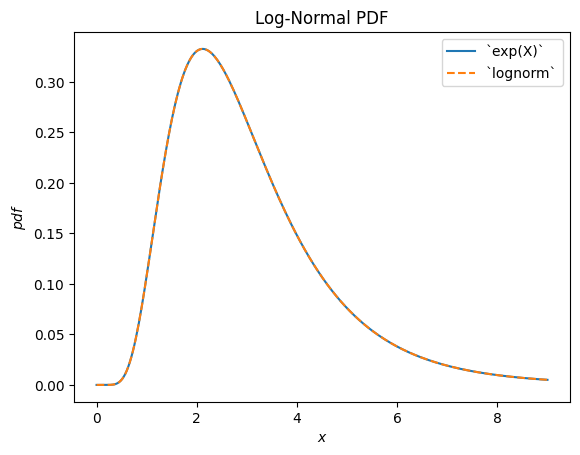
scipy.stats.lognormal 描述了常態分佈隨機變數的指數。之所以這樣命名,是因為所得隨機變數的對數呈常態分佈。(一般來說,「對數」分佈通常是隨機變數指數的基礎分佈。)
u, s = 1, 0.5
X = stats.Normal()*s + u
Y = stats.exp(X) # compare to `lognorm`
Y.plot(t=('x', eps, 9))
x = np.linspace(eps, 9, 300)
plt.plot(x, stats.lognorm(s=s, scale=np.exp(u)).pdf(x), '--')
plt.legend(['`exp(X)`', '`lognorm`'])
plt.title("Log-Normal PDF")
plt.show()
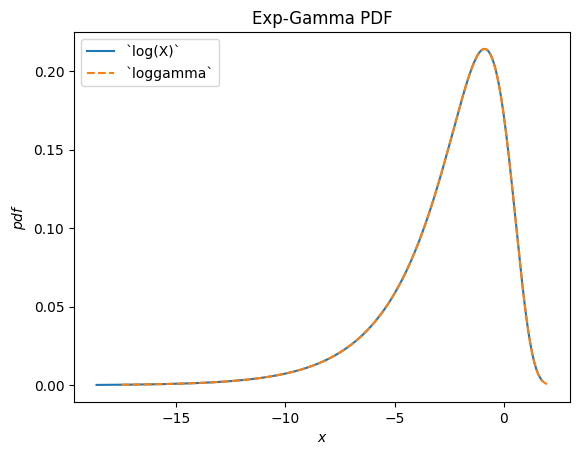
scipy.stats.loggamma 描述了 Gamma 分佈隨機變數的對數。請注意,根據典型的慣例,此分佈的適當名稱應為 exp-gamma,因為 RV 的指數呈 gamma 分佈。
Gamma = stats.make_distribution(stats.gamma)
a = 0.414
X = Gamma(a=a)
Y = stats.log(X) # compare to `loggamma`
Y.plot()
x = np.linspace(-17.5, 2, 300)
plt.plot(x, stats.loggamma(c=a).pdf(x), '--')
plt.legend(['`log(X)`', '`loggamma`'])
plt.title("Exp-Gamma PDF")
plt.show()
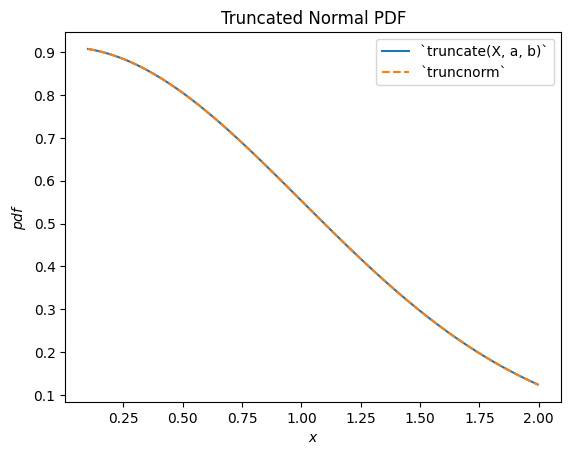
scipy.stats.truncnorm 是截斷常態隨機變數的基礎分佈。請注意,截斷轉換可以在平移和縮放常態分佈隨機變數之前或之後應用,這可以比 scipy.stats.truncnorm 更容易實現所需的結果(scipy.stats.truncnorm 本質上在截斷之後進行平移和縮放)。
a, b = 0.1, 2
X = stats.Normal()
Y = stats.truncate(X, a, b) # compare to `truncnorm`
Y.plot()
x = np.linspace(a, b, 300)
plt.plot(x, stats.truncnorm(a, b).pdf(x), '--')
plt.legend(['`truncate(X, a, b)`', '`truncnorm`'])
plt.title("Truncated Normal PDF")
plt.show()
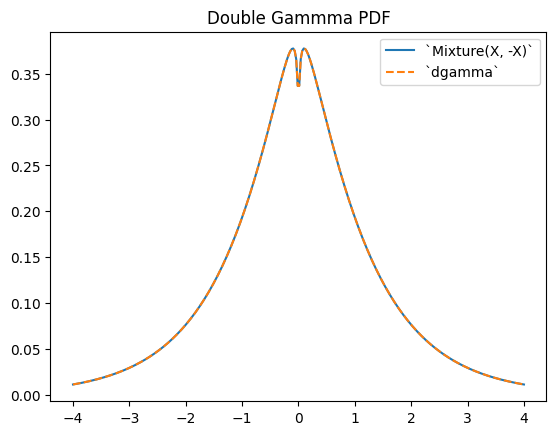
scipy.stats.dgamma 是兩個 gamma 分佈 RV 的混合,其中一個關於原點反射。(通常,「雙」分佈是 RV 混合的基礎分佈,其中一個被反射。)
a = 1.1
X = Gamma(a=a)
Y = stats.Mixture((X, -X), weights=[0.5, 0.5])
# plot method not available for mixtures
x = np.linspace(-4, 4, 300)
plt.plot(x, Y.pdf(x))
plt.plot(x, stats.dgamma(a=a).pdf(x), '--')
plt.legend(['`Mixture(X, -X)`', '`dgamma`'])
plt.title("Double Gammma PDF")
plt.show()
限制#
雖然支援隨機變數與 Python 純量或 NumPy 陣列之間的大多數算術轉換,但存在一些限制。
僅當引數為正整數時,才實作將隨機變數提升為引數的冪次。
僅當引數是 1 以外的正純量時,才實作將引數提升為隨機變數的冪次。
僅當支持完全是非負數或非正數時,才實作除以隨機變數。
僅當支持非負數時,才實作隨機變數的對數。(負值的對數是虛數。)
尚不支援兩個隨機變數之間的算術運算。請注意,此類運算在數學上要複雜得多;例如,兩個隨機變數之和的 PDF 涉及兩個 PDF 的卷積。
此外,雖然轉換是可組合的,但 a) 截斷和 b) 平移/縮放都只能執行一次。例如,Y = 2 * stats.exp(X + 1) 將引發錯誤,因為這將需要在指數運算之前進行平移,並在指數運算之後進行縮放;這被視為「平移/縮放」兩次。但是,這裡(以及在許多情況下)可以進行數學簡化以避免問題:Y = (2*math.exp(2)) * stats.exp(X) 是等效的,並且僅需要一次縮放運算。
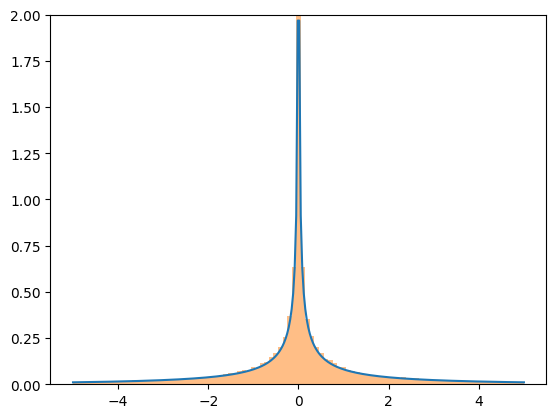
雖然轉換相當穩健,但它們都依賴於可能導致數值困難的通用實作。如果您擔心轉換結果的準確性,請考慮將結果 PDF 與隨機樣本的直方圖進行比較。
X = stats.Normal()
Y = X**3
x = np.linspace(-5, 5, 300)
plt.plot(x, Y.pdf(x), label='pdf')
plt.hist(X.sample(100000)**3, density=True, bins=np.linspace(-5, 5, 100), alpha=0.5);
plt.ylim(0, 2)
plt.show()
準蒙地卡羅抽樣#
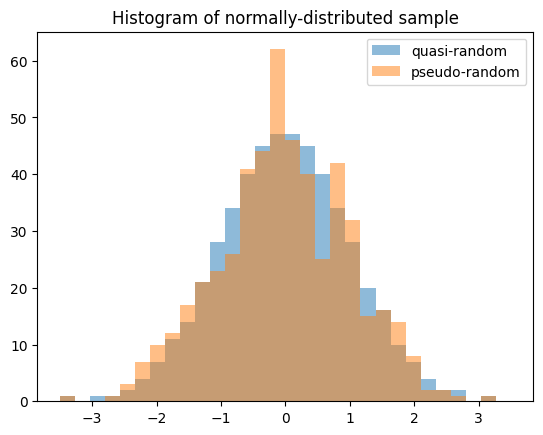
隨機變數可以從統計分佈中產生準隨機、低差異樣本。
import numpy as np
import matplotlib.pyplot as plt
from scipy import stats
X = stats.Normal()
rng = np.random.default_rng(7824387278234)
qrng = stats.qmc.Sobol(1, rng=rng) # instantiate a QMCEngine
bins = np.linspace(-3.5, 3.5, 31)
plt.hist(X.sample(512, rng=qrng), bins, alpha=0.5, label='quasi-random')
plt.hist(X.sample(512, rng=rng), bins, alpha=0.5, label='pseudo-random')
plt.title('Histogram of normally-distributed sample')
plt.legend()
plt.show()
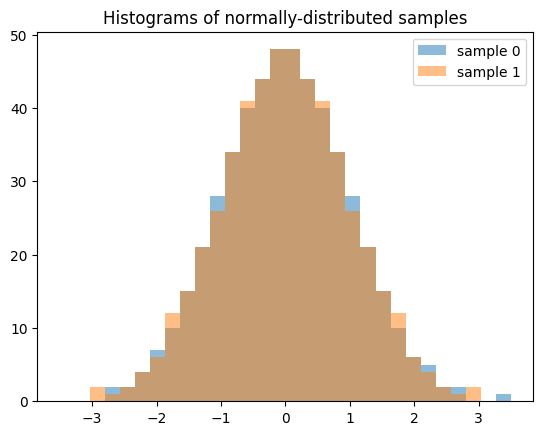
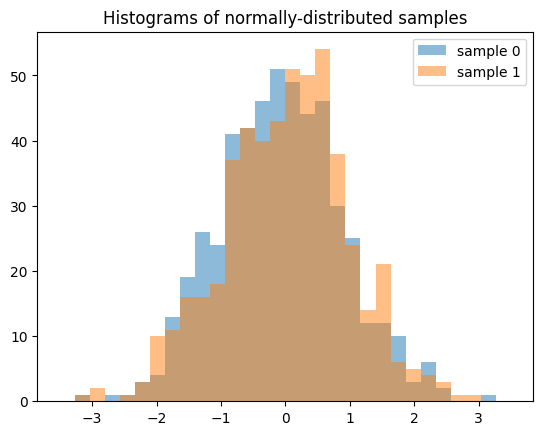
請注意,當產生多個樣本(例如 len(shape) > 1)時,沿著第零軸的每個切片都是獨立的低差異序列。以下產生兩個獨立的 QMC 樣本,每個樣本的長度為 512。
samples = X.sample((512, 2), rng=qrng)
plt.hist(samples[:, 0], bins, alpha=0.5, label='sample 0')
plt.hist(samples[:, 1], bins, alpha=0.5, label='sample 1')
plt.title('Histograms of normally-distributed samples')
plt.legend()
plt.show()
如果我們產生 512 個獨立的 QMC 序列,每個序列的長度為 2,則結果會大不相同。
samples = X.sample((2, 512), rng=qrng)
plt.hist(samples[0], bins, alpha=0.5, label='sample 0')
plt.hist(samples[1], bins, alpha=0.5, label='sample 1')
plt.title('Histograms of normally-distributed samples')
plt.legend()
plt.show()
準確性#
對於某些分佈,例如 scipy.stats.norm,幾乎所有分佈的方法都經過自訂以確保準確的計算。對於其他分佈,例如 scipy.stats.gausshyper,除了 PDF 之外,幾乎沒有定義其他內容,並且其他方法必須根據 PDF 進行數值計算。例如,Gauss 超幾何分佈的生存函數(互補 CDF)是透過將 PDF 從 0(支持的左端)數值積分到 x 以獲得 CDF,然後取補數來計算的。
a, b, c, z = 1.5, 2.5, 2, 0
x = 0.5
frozen = stats.gausshyper(a=a, b=b, c=c, z=z)
frozen.sf(x)
0.28779340921080643
frozen.sf(x) == 1 - integrate.quad(frozen.pdf, 0, x)[0]
True
但是,另一種方法是將 PDF 從 x 數值積分到 1(支持的右端)。
integrate.quad(frozen.pdf, x, 1)[0]
0.28779340919669805
這些是不同但同樣有效的方法,因此假設 PDF 是準確的,則兩個結果不太可能以相同的方式不準確。因此,我們可以透過比較它們來估計結果的準確性。
res1 = frozen.sf(x)
res2 = integrate.quad(frozen.pdf, x, 1)[0]
abs((res1 - res2) / res1)
4.902259981810363e-11
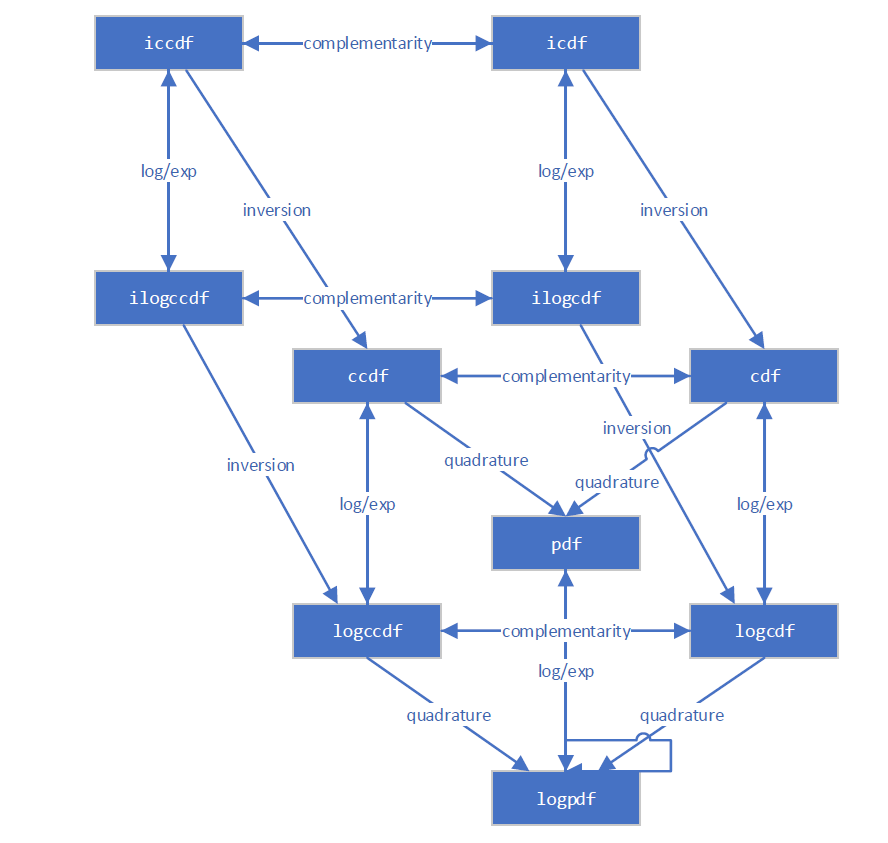
新的基礎架構意識到計算大多數量的幾種不同方法。例如,此圖說明了各種分佈函數之間的關係。
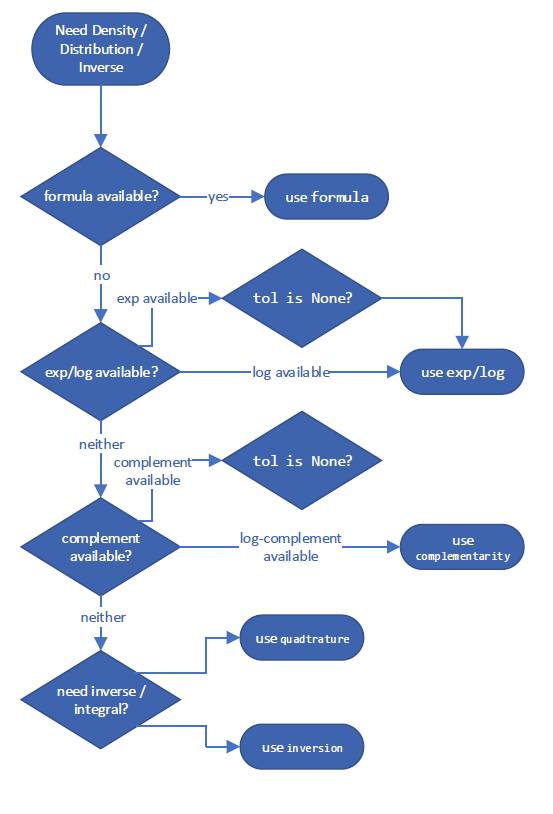
它遵循決策樹來選擇預期最準確的估計量的方法。這些用於「最佳」方法的特定決策樹可能會在 SciPy 版本之間發生變化,但用於計算互補 CDF 的範例可能如下所示
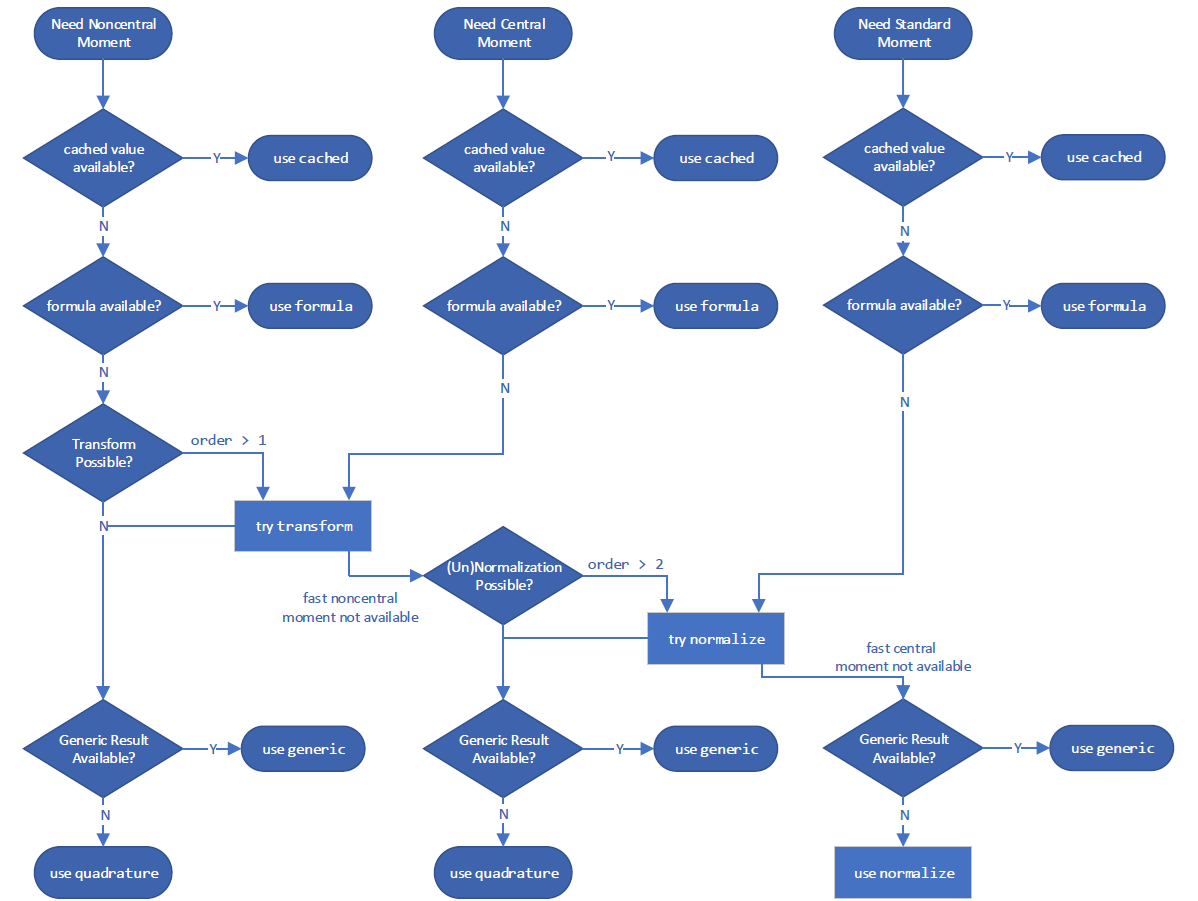
以及用於計算動差
但是,您可以使用 method 引數覆寫它使用的方法。
GaussHyper = stats.make_distribution(stats.gausshyper)
X = GaussHyper(a=a, b=b, c=c, z=z)
對於 ccdf,method='quadrature' 在右尾積分 PDF。
X.ccdf(x, method='quadrature') == integrate.tanhsinh(X.pdf, x, 1).integral
True
method='complement' 取 CDF 的補數。
X.ccdf(x, method='complement') == (1 - integrate.tanhsinh(X.pdf, 0, x).integral)
True
method='logexp' 取 CCDF 對數的指數。
X.ccdf(x, method='logexp') == np.exp(integrate.tanhsinh(lambda x: X.logpdf(x), x, 1, log=True).integral)
True
如有疑問,請考慮嘗試不同的 method 來計算給定的量。
效能#
考慮涉及 Gauss 超幾何分佈的計算效能。對於純量形狀和引數,新舊基礎架構的效能相當。新的基礎架構預計不會快多少;雖然它減少了參數驗證的「開銷」,但對於純量量的數值積分而言,它並沒有顯著加快速度,而後者在這裡佔據了主要的執行時間。
%timeit X.cdf(x) # new infrastructure
402 µs ± 868 ns per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
%timeit stats.gausshyper.cdf(x, a, b, c, z) # old infrastructure
392 µs ± 955 ns per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
np.isclose(X.cdf(x), stats.gausshyper.cdf(x, a, b, c, z))
True
但是,當涉及陣列時,新的基礎架構要快得多。這是因為為新基礎架構開發的底層積分器 (scipy.integrate.tanhsinh) 和求根器 (scipy.optimize.elementwise.find_root) 本身是向量化的,而舊基礎架構使用的舊例程 (scipy.integrate.quad 和 scipy.optimize.brentq) 不是。
x = np.linspace(0, 1, 1000)
%timeit X.cdf(x) # new infrastructure
2.47 ms ± 47.6 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
%timeit stats.gausshyper.cdf(x, a, b, c, z) # old infrastructure
330 ms ± 150 µs per loop (mean ± std. dev. of 7 runs, 1 loop each)
對於每次底層函數透過數值求積或反演(求根)計算的陣列計算,都可以預期獲得相當的效能提升。
視覺化#
我們可以使用便利的方法 plot,輕鬆地視覺化隨機變數底層分布的函數。
import matplotlib.pyplot as plt
ax = X.plot()
plt.show()
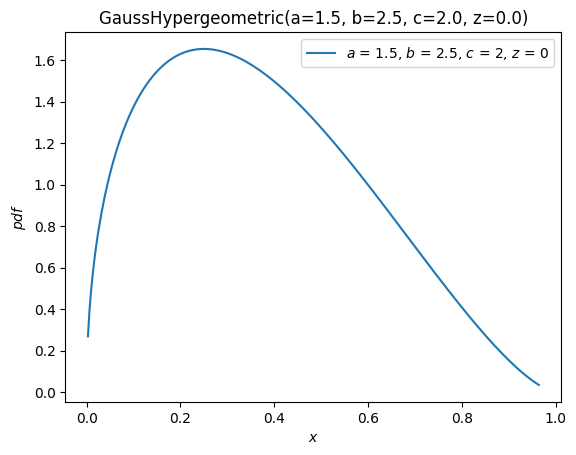
X.plot(y='cdf')
plt.show()
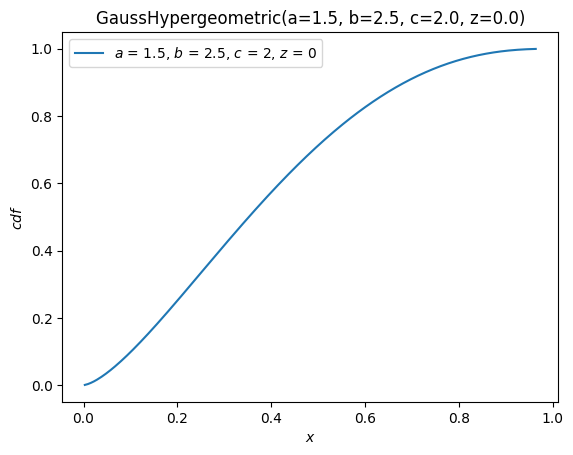
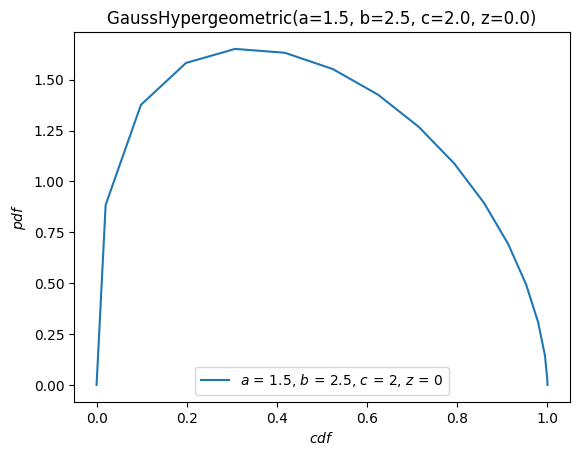
plot 方法非常彈性,其簽名受到 The Grammar of Graphics 的啟發。例如,對於在 \(x\) 範圍 \([-10, 10]\) 內的參數,繪製 PDF 對 CDF 的圖。
X.plot('cdf', 'pdf', t=('x', -10, 10))
plt.show()
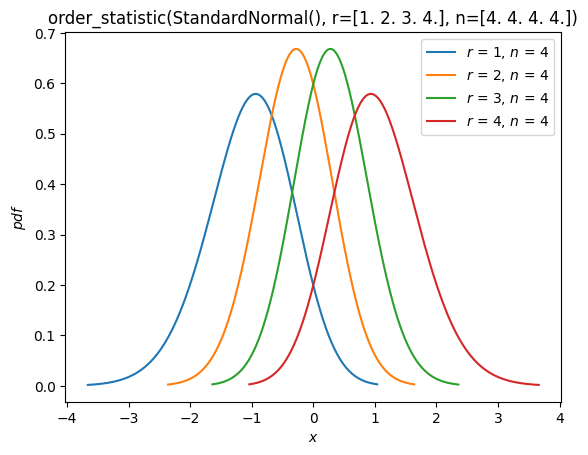
順序統計量#
如同在上面的「擬合」章節中所見,現在已支援來自分布之隨機樣本的順序統計量分布。例如,我們可以繪製樣本大小為 4 之常態分布的順序統計量的機率密度函數。
n = 4
r = np.arange(1, n+1)
X = stats.Normal()
Y = stats.order_statistic(X, r=r, n=n)
Y.plot()
plt.show()
計算這些順序統計量的期望值
Y.mean()
array([-1.02937537, -0.29701138, 0.29701138, 1.02937537])
結論#
儘管改變可能令人感到不適,新的基礎架構為改善 SciPy 概率分布用戶的體驗鋪平了道路。當然,它還不完善!在本指南撰寫之時,以下功能計劃在未來版本中推出
額外的內建分布(除了
Normal和Uniform之外)離散分布
循環分布,包括將任意連續分布環繞在圓上的能力
支援 NumPy 以外的其他 Python Array API 相容後端(例如 CuPy、PyTorch、JAX)
一如既往,SciPy 的開發是為了 SciPy 社群,並由 SciPy 社群完成。我們始終感謝您對其他功能的建議以及實作方面的幫助。
我們希望本指南能激發您嘗試新基礎架構的優勢,並簡化流程。