Kendall tau 檢定#
Kendall tau 是衡量兩個排序之間對應關係的指標。
考慮以下來自 [1] 的資料,該研究探討了不健康人體肝臟中游離脯胺酸(一種胺基酸)和總膠原蛋白(一種常見於結締組織的蛋白質)之間的關係。
下方的 x 和 y 陣列記錄了這兩種化合物的測量值。這些觀察結果是成對的:每個游離脯胺酸的測量值都與在相同索引處測量總膠原蛋白的肝臟相同。
import numpy as np
# total collagen (mg/g dry weight of liver)
x = np.array([7.1, 7.1, 7.2, 8.3, 9.4, 10.5, 11.4])
# free proline (μ mole/g dry weight of liver)
y = np.array([2.8, 2.9, 2.8, 2.6, 3.5, 4.6, 5.0])
這些資料在 [2] 中使用 Spearman 相關係數進行了分析,Spearman 相關係數是一種與 Kendall tau 相似的統計量,因為它也對樣本之間的順序相關性敏感。讓我們使用 Kendall's tau 進行類似的研究。
from scipy import stats
res = stats.kendalltau(x, y)
res.statistic
0.5499999999999999
對於具有強烈正向順序相關性的樣本,此統計量的值往往很高(接近 1);對於具有強烈負向順序相關性的樣本,此統計量的值往往很低(接近 -1);而對於具有弱順序相關性的樣本,此統計量的值幅度很小(接近於零)。
此檢定是通過將觀察到的統計量值與虛無分佈進行比較來執行的:虛無分佈是在總膠原蛋白和游離脯胺酸測量值獨立的虛無假設下得出的統計量值的分佈。
對於此檢定,無領結的大樣本的虛無分佈近似為常態分佈,變異數為 (2*(2*n + 5))/(9*n*(n - 1)),其中 n = len(x)。
import matplotlib.pyplot as plt
n = len(x) # len(x) == len(y)
var = (2*(2*n + 5))/(9*n*(n - 1))
dist = stats.norm(scale=np.sqrt(var))
z_vals = np.linspace(-1.25, 1.25, 100)
pdf = dist.pdf(z_vals)
fig, ax = plt.subplots(figsize=(8, 5))
def plot(ax): # we'll reuse this
ax.plot(z_vals, pdf)
ax.set_title("Kendall Tau Test Null Distribution")
ax.set_xlabel("statistic")
ax.set_ylabel("probability density")
plot(ax)
plt.show()
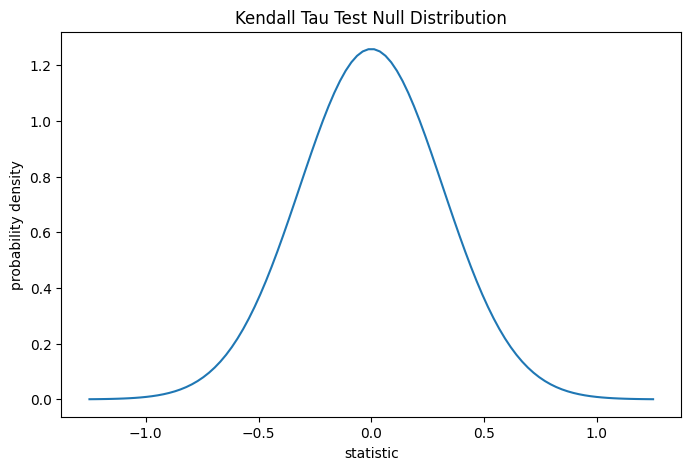
此比較由 p 值量化:p 值是虛無分佈中與觀察到的統計量值一樣極端或更極端的值的比例。在統計量為正值的雙尾檢定中,虛無分佈中大於轉換後統計量的元素和虛無分佈中小於觀察到的統計量的負值的元素都被視為「更極端」。
fig, ax = plt.subplots(figsize=(8, 5))
plot(ax)
pvalue = dist.cdf(-res.statistic) + dist.sf(res.statistic)
annotation = (f'p-value={pvalue:.4f}\n(shaded area)')
props = dict(facecolor='black', width=1, headwidth=5, headlength=8)
_ = ax.annotate(annotation, (0.65, 0.15), (0.8, 0.3), arrowprops=props)
i = z_vals >= res.statistic
ax.fill_between(z_vals[i], y1=0, y2=pdf[i], color='C0')
i = z_vals <= -res.statistic
ax.fill_between(z_vals[i], y1=0, y2=pdf[i], color='C0')
ax.set_xlim(-1.25, 1.25)
ax.set_ylim(0, 0.5)
plt.show()
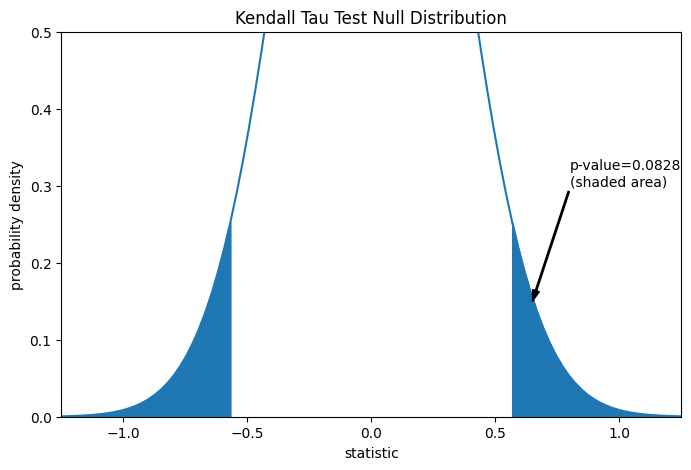
res.pvalue
0.09108705741631495
請注意,曲線的陰影區域與 scipy.stats.kendalltau 返回的 p 值之間存在輕微差異。這是因為我們的資料有領結,並且我們忽略了 scipy.stats.kendalltau 執行的對虛無分佈變異數的領結校正。對於沒有領結的樣本,我們圖表的陰影區域和 scipy.stats.kendalltau 返回的 p 值將完全匹配。
如果 p 值「很小」——也就是說,如果從獨立分佈中採樣資料產生如此極端的統計量值的機率很低——這可以被視為反對虛無假設而支持備擇假設的證據:總膠原蛋白和游離脯胺酸的分佈不是獨立的。請注意
反之則不然;也就是說,此檢定不用於為虛無假設提供證據。
將被視為「小」的值的閾值是一個選擇,應在分析資料 [3] 之前做出,並考慮到偽陽性(錯誤地拒絕虛無假設)和偽陰性(未能拒絕錯誤的虛無假設)的風險。
小的 p 值不是大型效應的證據;相反,它們只能為「顯著」效應提供證據,這意味著它們不太可能在虛無假設下發生。
對於中等大小的無領結樣本,scipy.stats.kendalltau 可以精確計算 p 值。但是,在存在領結的情況下,scipy.stats.kendalltau 會採用漸近近似。儘管如此,我們可以使用置換檢定來精確計算虛無分佈:在總膠原蛋白和游離脯胺酸獨立的虛無假設下,每個游離脯胺酸測量值都同樣有可能與任何總膠原蛋白測量值一起觀察到。因此,我們可以通過計算 x 和 y 之間每種可能的元素配對下的統計量來形成精確的虛無分佈。
def statistic(x): # explore all possible pairings by permuting `x`
return stats.kendalltau(x, y).statistic # ignore pvalue
ref = stats.permutation_test((x,), statistic,
permutation_type='pairings')
fig, ax = plt.subplots(figsize=(8, 5))
plot(ax)
bins = np.linspace(-1.25, 1.25, 25)
ax.hist(ref.null_distribution, bins=bins, density=True)
ax.legend(['asymptotic approximation\n(many observations)',
'exact null distribution'])
plot(ax)
plt.show()
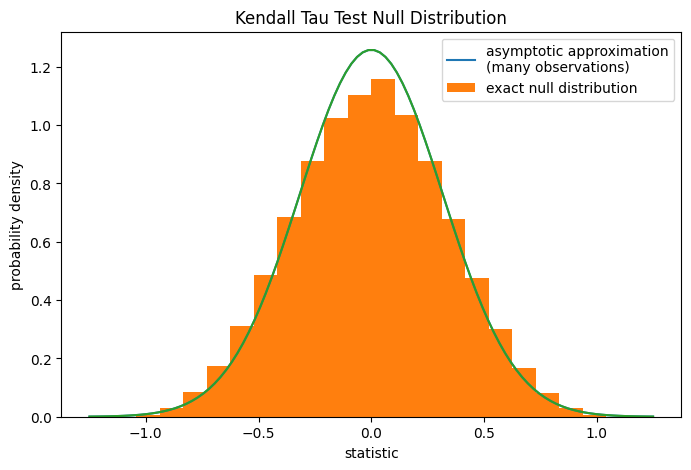
ref.pvalue
0.12222222222222222
請注意,此處計算的精確 p 值與上方 scipy.stats.kendalltau 返回的近似值之間存在顯著差異。對於有領結的小樣本,請考慮執行置換檢定以獲得更準確的結果。