核密度估計#
統計學中常見的任務是從一組資料樣本估計隨機變數的機率密度函數 (PDF)。此任務稱為密度估計。最廣為人知的工具是直方圖。直方圖是視覺化的實用工具(主要是因為每個人都理解它),但沒有非常有效地利用可用的資料。核密度估計 (KDE) 是用於相同任務的更有效工具。scipy.stats.gaussian_kde 估計器可用於估計單變數以及多變數資料的 PDF。如果資料是單峰的,效果最佳。
單變數估計#
我們從最少量的資料開始,以了解 scipy.stats.gaussian_kde 的運作方式,以及頻寬選擇的不同選項的作用。從 PDF 中抽樣的資料在圖的底部顯示為藍色短劃線(這稱為地毯圖)
>>> import numpy as np
>>> from scipy import stats
>>> import matplotlib.pyplot as plt
>>> x1 = np.array([-7, -5, 1, 4, 5], dtype=np.float64)
>>> kde1 = stats.gaussian_kde(x1)
>>> kde2 = stats.gaussian_kde(x1, bw_method='silverman')
>>> fig = plt.figure()
>>> ax = fig.add_subplot(111)
>>> ax.plot(x1, np.zeros(x1.shape), 'b+', ms=20) # rug plot
>>> x_eval = np.linspace(-10, 10, num=200)
>>> ax.plot(x_eval, kde1(x_eval), 'k-', label="Scott's Rule")
>>> ax.plot(x_eval, kde2(x_eval), 'r-', label="Silverman's Rule")
>>> plt.show()
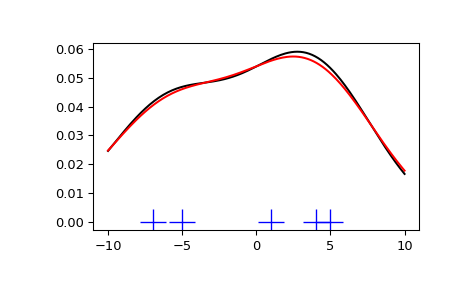
我們看到 Scott 規則和 Silverman 規則之間幾乎沒有差異,並且使用有限量資料的頻寬選擇可能有點太寬。我們可以定義自己的頻寬函數以獲得較不平滑的結果。
>>> def my_kde_bandwidth(obj, fac=1./5):
... """We use Scott's Rule, multiplied by a constant factor."""
... return np.power(obj.n, -1./(obj.d+4)) * fac
>>> fig = plt.figure()
>>> ax = fig.add_subplot(111)
>>> ax.plot(x1, np.zeros(x1.shape), 'b+', ms=20) # rug plot
>>> kde3 = stats.gaussian_kde(x1, bw_method=my_kde_bandwidth)
>>> ax.plot(x_eval, kde3(x_eval), 'g-', label="With smaller BW")
>>> plt.show()
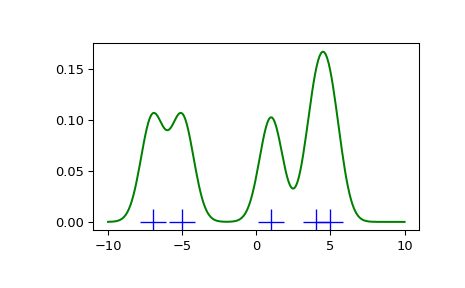
我們看到,如果我們將頻寬設定為非常窄,則獲得的機率密度函數 (PDF) 估計值只是每個資料點周圍高斯函數的總和。
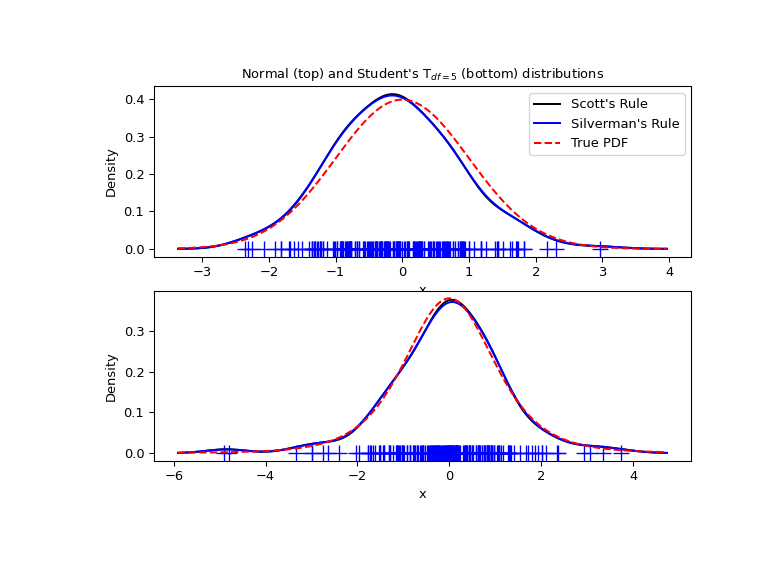
我們現在來看一個更實際的例子,並查看兩個可用的頻寬選擇規則之間的差異。已知這些規則對於(接近)常態分佈效果良好,但即使對於非常強烈非常態的單峰分佈,它們也能合理地良好地運作。作為非常態分佈,我們採用自由度為 5 的 Student's T 分佈。
import numpy as np
import matplotlib.pyplot as plt
from scipy import stats
rng = np.random.default_rng()
x1 = rng.normal(size=200) # random data, normal distribution
xs = np.linspace(x1.min()-1, x1.max()+1, 200)
kde1 = stats.gaussian_kde(x1)
kde2 = stats.gaussian_kde(x1, bw_method='silverman')
fig = plt.figure(figsize=(8, 6))
ax1 = fig.add_subplot(211)
ax1.plot(x1, np.zeros(x1.shape), 'b+', ms=12) # rug plot
ax1.plot(xs, kde1(xs), 'k-', label="Scott's Rule")
ax1.plot(xs, kde2(xs), 'b-', label="Silverman's Rule")
ax1.plot(xs, stats.norm.pdf(xs), 'r--', label="True PDF")
ax1.set_xlabel('x')
ax1.set_ylabel('Density')
ax1.set_title("Normal (top) and Student's T$_{df=5}$ (bottom) distributions")
ax1.legend(loc=1)
x2 = stats.t.rvs(5, size=200, random_state=rng) # random data, T distribution
xs = np.linspace(x2.min() - 1, x2.max() + 1, 200)
kde3 = stats.gaussian_kde(x2)
kde4 = stats.gaussian_kde(x2, bw_method='silverman')
ax2 = fig.add_subplot(212)
ax2.plot(x2, np.zeros(x2.shape), 'b+', ms=12) # rug plot
ax2.plot(xs, kde3(xs), 'k-', label="Scott's Rule")
ax2.plot(xs, kde4(xs), 'b-', label="Silverman's Rule")
ax2.plot(xs, stats.t.pdf(xs, 5), 'r--', label="True PDF")
ax2.set_xlabel('x')
ax2.set_ylabel('Density')
plt.show()
我們現在看看雙峰分佈,其中一個較寬,一個較窄的高斯特徵。我們預期這將是一個更難以近似的密度,因為準確解析每個特徵需要不同的頻寬。
>>> from functools import partial
>>> loc1, scale1, size1 = (-2, 1, 175)
>>> loc2, scale2, size2 = (2, 0.2, 50)
>>> x2 = np.concatenate([np.random.normal(loc=loc1, scale=scale1, size=size1),
... np.random.normal(loc=loc2, scale=scale2, size=size2)])
>>> x_eval = np.linspace(x2.min() - 1, x2.max() + 1, 500)
>>> kde = stats.gaussian_kde(x2)
>>> kde2 = stats.gaussian_kde(x2, bw_method='silverman')
>>> kde3 = stats.gaussian_kde(x2, bw_method=partial(my_kde_bandwidth, fac=0.2))
>>> kde4 = stats.gaussian_kde(x2, bw_method=partial(my_kde_bandwidth, fac=0.5))
>>> pdf = stats.norm.pdf
>>> bimodal_pdf = pdf(x_eval, loc=loc1, scale=scale1) * float(size1) / x2.size + \
... pdf(x_eval, loc=loc2, scale=scale2) * float(size2) / x2.size
>>> fig = plt.figure(figsize=(8, 6))
>>> ax = fig.add_subplot(111)
>>> ax.plot(x2, np.zeros(x2.shape), 'b+', ms=12)
>>> ax.plot(x_eval, kde(x_eval), 'k-', label="Scott's Rule")
>>> ax.plot(x_eval, kde2(x_eval), 'b-', label="Silverman's Rule")
>>> ax.plot(x_eval, kde3(x_eval), 'g-', label="Scott * 0.2")
>>> ax.plot(x_eval, kde4(x_eval), 'c-', label="Scott * 0.5")
>>> ax.plot(x_eval, bimodal_pdf, 'r--', label="Actual PDF")
>>> ax.set_xlim([x_eval.min(), x_eval.max()])
>>> ax.legend(loc=2)
>>> ax.set_xlabel('x')
>>> ax.set_ylabel('Density')
>>> plt.show()
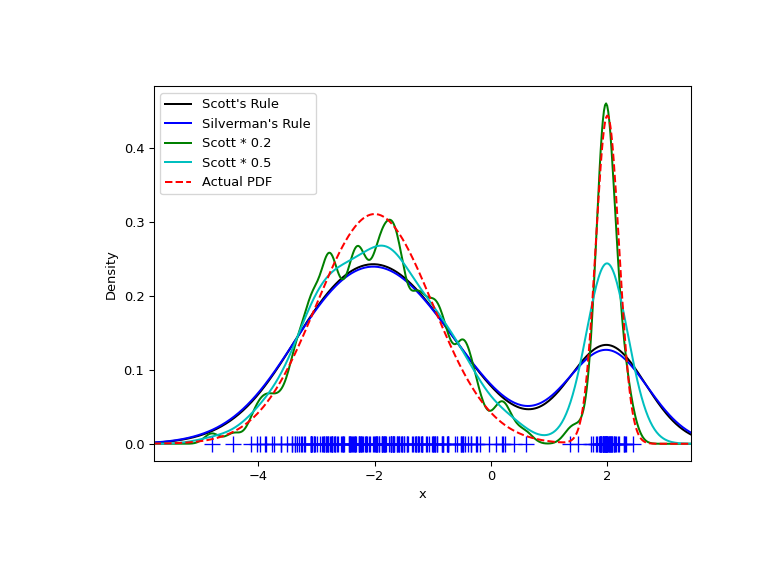
正如預期的那樣,由於雙峰分佈的兩個特徵具有不同的特徵尺寸,KDE 並不像我們希望的那樣接近真實 PDF。透過將預設頻寬減半(Scott * 0.5),我們可以做得更好一些,而使用比預設值小 5 倍的頻寬則不夠平滑。但是,在這種情況下,我們真正需要的是非均勻(自適應)頻寬。
多變數估計#
使用 scipy.stats.gaussian_kde,我們可以執行多變數以及單變數估計。我們示範二變數的情況。首先,我們使用兩個變數相關的模型生成一些隨機資料。
>>> def measure(n):
... """Measurement model, return two coupled measurements."""
... m1 = np.random.normal(size=n)
... m2 = np.random.normal(scale=0.5, size=n)
... return m1+m2, m1-m2
>>> m1, m2 = measure(2000)
>>> xmin = m1.min()
>>> xmax = m1.max()
>>> ymin = m2.min()
>>> ymax = m2.max()
然後我們將 KDE 應用於資料
>>> X, Y = np.mgrid[xmin:xmax:100j, ymin:ymax:100j]
>>> positions = np.vstack([X.ravel(), Y.ravel()])
>>> values = np.vstack([m1, m2])
>>> kernel = stats.gaussian_kde(values)
>>> Z = np.reshape(kernel.evaluate(positions).T, X.shape)
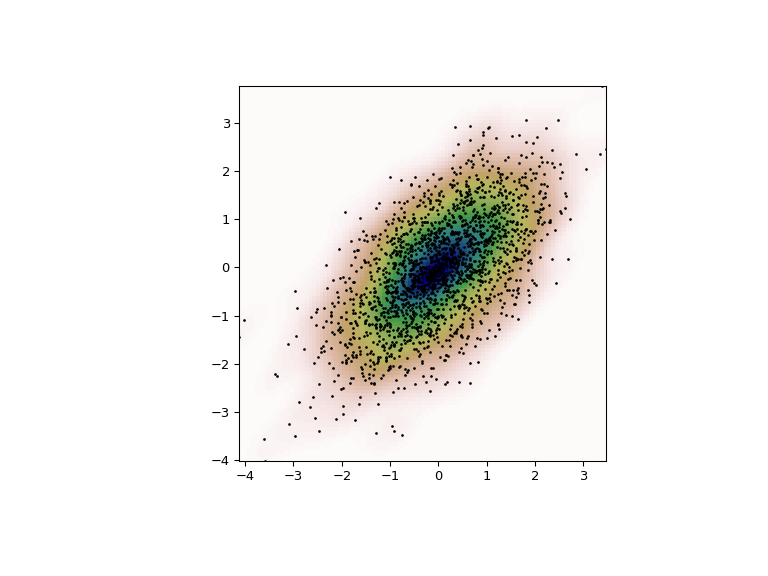
最後,我們將估計的二變數分佈繪製為色譜圖,並在頂部繪製個別資料點。
>>> fig = plt.figure(figsize=(8, 6))
>>> ax = fig.add_subplot(111)
>>> ax.imshow(np.rot90(Z), cmap=plt.cm.gist_earth_r,
... extent=[xmin, xmax, ymin, ymax])
>>> ax.plot(m1, m2, 'k.', markersize=2)
>>> ax.set_xlim([xmin, xmax])
>>> ax.set_ylim([ymin, ymax])
>>> plt.show()